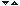 | Biologie | Chimie | Didactica | Fizica | Geografie | Informatica |
| Istorie | Literatura | Matematica | Psihologie |
RAPORT DE CERCETARE (RC)
EVALUAREA DISTRIBUTIEI SI CARACTERIZAREA VARIABILITATII GENETICE LA SALMONIDELE DIN ROMANIA
Etapa III/2010
Identificarea si distributia salmonidelor pe bazine hidrografice. Optimizarea protocoalelor de amplificare a regiunii D-loop ADNmt.
Activitatea III.1: Stabilirea bazinelor hidrografice cu prezenta permanenta sau sporadica a salmonidelor.
Activitatea III.2: Culegerea de date si recoltarea, transportul si conservarea de probe biologice.
Activitatea III.3: Analize fizico-chimice privind calitatea apei habitatelor studiate.
Activitate III.4: Identificarea populatiilor autohtone de salmonide pe bazine hidrografice.
Activitatea III.5: Optimizarea protocoalelor PCR, PCR-RFLP si de secvetiere pentru regiunea D-loop ADNmt.
Activitatea III.6: Secventierea si introducerea in Genome DataBase a secventelor noi obtinute.
Activitatea III.7: Identificarea factorilor limitativi ai habitatelor si implicit ai cresteri salmonidelor.
Activitatea III.8: Editarea unei harti privind distributia salmonidelor pe bazine hidrografice.
Activitatea III.9: Analiza comparativa cu secventele ADN din regiunea D-Loop mitocondriala existente in Genome Data Base.
Activitatea III.10: Diseminarea rezultatelor prin publicarea acestora in reviste internationale si participarea la conferinte.
Activitatea III.11: Completare pagina web cu informatii despre secventele mitocondriale studiate.
REZUMAT
Salmonidele reprezinta un grup heterogen de pesti reuniti intr-o singura familie, Salmonidae, cu 66 de specii. In fauna tarii noastre sunt prezente la ora actuala 6 specii de salmonide: Salmo trutta fario (pastravul indigen), Salmo trutta labrax (pastravul de Marea Neagra), Salvellinus fontinallis (fantanelul), Hucho hucho (lostrita), Thymallus thymallus (lipanul) si Onchorynchus mykiss (pastravul curcubeu).
In cadrul acestei etape a fost realizata o trecere in revista a situatiei calitatii apei Dunarii, principalul habitat de reproducere si crestere a puilor si juvenililor de pastrav de Marea Neagra, pentru perioada 2005 - 2009, mentionandu-se valorile de pH si alti indicatori de poluare cu metale, nitriti, nitrati si alti compusi chimici. De asemenea, s-a realizat o evaluare a calitatii apei si pentru bazinele hidrografice populate cu salmonide din zona Munteniei, intocmindu-se si harti care cuprind distributia acestor specii de pesti pe bazine hidrografice.
In ciuda importantei salmonidelor, istoria lor evolutiva a constituit, pentru o lunga perioada de timp, un subiect de disputa. Speciile de salmonide din Romania au fost caracterizate doar din punct de vedere taxonomic prin metode clasice: analize biometrice si morfologice. Tinand seama de importanta economica si stiintifica a acestor specii, este absolut necesara realizarea la salmonide a unor studii de filogenie moleculara bazate pe analiza ADN nuclear si mitocondrial. Intre genele ARNtPro si ARNtPhe din genomul mitocondrial al vertebratelor se gaseste o regiune necodificatoare care variaza ca lungime. Regiunea mitocondriala de control (D-Loop) reprezinta regiunea reglatoare a genomului mitocondrial al vertebratelor, implicata in initierea replicarii ADN.
In vederea analizei filogenetice, pe baza regiunii mitocondriale D-loop, s-a determinat initial temperatura de hibridizare optima pentru fiecare set de primeri prin PCR in gradient de temperatura. In final a fost optimizat protocolul de amplificare pentru regiunea control D-loop si pentru regiunea mitocondriala extinsa la speciile de salmonide analizate si au fost alese temperaturile optime pentru hibridizarea primerilor.
In urma analizelor PCR-RFLP s-a observat ca scindarea ampliconilor cu endonucleaza de restrictie Hinf I, urmata de analiza produsilor rezultati in gel de agaroza conduce la identificarea diferentelor dintre diferitele specii de salmonide. Tehnica pusa astfel la punct poate fi utilizata cu succes in identificarea moleculara a diferitelor specii de salmonide si poate fi testata si pentru analize moleculare ale hibrizilor dintre diferite specii.
Fragmentele obtinute pentru regiunea de control mitocondriala (D-loop) la speciile de salmonide din Romania au fost secventiate si comparate cu secvente similare din baza de date GenBank. Prin metodele NJ si Maximum Parcimony (MP) au fost construiti arbori care ilustreaza relatiile filogenetice intre cele 17 specii de salmonide analizate. Arborii obtinuti prin cele doua metode prezinta o topologie similara, singura diferenta fiind dispunerea genului Salvelinus in raport cu genurile Oncorhynchus si Salmo. La baza arborilor filogenetici sunt dispuse speciile primitive de salmonide, cum ar fi Thymallus thymallus, Thymallus articus si Coregonus lavaraetus, apartinand subafmiliilor Thymallinae si Coregoninae, subafamilii care au aparut inaintea subfamiliei Salmoninae. Subfamilia Salmoninae este alcatuita din genurile Salmo, Oncorhynchus, Hucho, Brachymistax si Salvelinus, iar, conform analizei noastre, acestea formeaza grupuri monofiletice. Dintre Salmoninae, cele mai primitive specii sunt cele apartinand genurilor Brachymistax si Hucho. Reprezentantii genului Salmo formeaza o clada distincta, in care Salmo salar sta la originea celorlate specii.
Speciile genului Oncorhynchus formeaza o clada distincta fata de celelalte grupuri de salmonide. Haplotipurile din Romania se incadreaza in grupul celorlalte haplotipuri de la O. mykiss. Speciile genului Salvelnius formeaza un grup monofiletic plasat in cadrul Salmoninaelor intre speciile genului Salmo si Onchorhynchus. Pozitia ocupata de Salvelinus contrazice ipoteza filogeniei clasice conform careia genurile Salmo si Oncorhynchus sunt cel mai apropiate din punct de vedere filogenetic si vin in sprijinul anumitor date de filogenie moleculara care afirma ca intre genurile Salvelinus si Oncorhynchus exista o apropiere filogenetica mai mare.
In cazul analizei noastre bazate pe secventa D-loop prin NJ sunt sustinute datele moleculare mentionate anterior. In schimb, metoda MP confirma ipoteza filogeniei clasice conform careia Salmo si Oncorhynchus reprezinta taxoni foarte apropiati din punct de vedere evolutiv.
CONSIDERATII GENERALE
Importanta salmonidelor se manifesta atat la nivel comercial, cat si social. Unele specii de salmonide sunt crescute in conditii intensive in pastravarii, pentru consum alimentar si sunt de asemenea, foarte cautate pentru pescuit sportiv.
Salmonidele reprezinta un grup heterogen de pesti reuniti intr-o singura familie, Salmonidae, cu 66 de specii. Majoritatea speciilor prefera in general apele reci, limpezi, cu fund pietros si bine oxigenate, in special din zona montana. Exceptie face Salmo trutta labrax, specie endemica bazinului Pontic, intalnita in Marea Neagra si Marea Azov si care isi desfasoara ciclul trofic in mediul marin. Romania este una dintre tarile europene cu un important potential salmonicol natural si cu bune perspective in dezvoltarea cresterii intensive a salmonidelor. In fauna tarii noastre sunt prezente la ora actuala 6 specii de salmonide: Salmo trutta fario (pastravul indigen), Salmo trutta labrax (pastravul de Marea Neagra), Salvellinus fontinallis (fantanelul), Hucho hucho (lostrita), Thymallus thymallus (lipanul) si Onchorynchus mykiss (pastravul curcubeu). La aceste specii se adauga reprezentanti ai genului Coregonus (speciile Coregonus albula ladogensis si Coregonus lavaraetus maraenoides) care au fost introdusi expeimental in anumite lacuri din Romania, dar despre care nu exista date privind adaptarea si dezvoltarea.
Pescuitul excesiv, construirea barajelor, poluarea si alti factori antropici care au determinat reducerea efectivelor de salmonide din mediul natural au impus adoptarea unor solutii de tipul dezvoltarii salmoniculturii intensive. In plus, pastravul ca si aliment de consum este perceput ca bun pentru sanatatea omului pentru ca este sarac in calorii, bogat in proteine, vitamine, fier, fosfor si lecitina. Acesta reprezinta unul dintre motivele pentru care cresterea salmonidelor in sistem intensiv a cunoscut o mare dezvoltare.
Salmonicultura moderna a avut crestere exploziva incepand cu anii '80 ai secolului trecut, in special in Marea Britanie si Danemarca. Obtinerea si cresterea salmonidelor in conditii intensive necesita ape reci, bogate in oxigen si cu un nivel foarte scazut de poluanti, departe de marile centre urbane. Desi, salmonidele sunt specii cu originea in emisfera nordica, aproximativ 40% din productia salmonicola provine din tari din emisfera sudica, unde aceste specii au fost introduse cu succes. Chile si Norvegia sunt lideri la productia de somon si pastrav, detinand aproximativ 76% din totalul pietei. Somonul de Atlantic (Salmo salar) este specia cel mai frecvent utilizata in acvacultura salmonidelor, urmata de pastravul curcubeu (Oncorhynchus mykiss) (Figura 1).
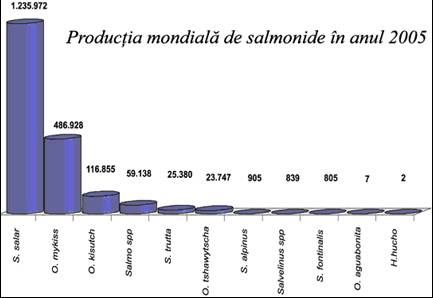
Figura 1. Principalele specii de salmonide utilizate in acvacultura. Cifrele sunt exprimate in tone. (Sursa: FAO global databases: Aquaculture production 2005).
In Romania, Regia Nationala a Padurilor gestioneaza 43 de pastravarii. La acestea se adauga un numar important de pastravarii din sectorul privat. La ora actuala, speciile folosite in cresterea intensiva la noi in tara sunt: pastravul indigen (Salmo trutta fario L.), pastravul fantanel (Salvelinus fontinalis M), pastravul curcubeu (Oncorhynchus mykiss W.) si lostrita (Hucho hucho L.). Dintre acestea pastravul curcubeu se preteaza cel mai bine la cresterea intensiva.
Totusi, la ora actuala, exista o practica destul de periculoasa pentru viitorul conservarii genofondului salmonicol din mediul natural: pentru a compensa declinul populational si suporta presiunea pescuitului sportiv se practica repopularea apelor de munte cu exemplare din specii neadaptate si hibrizi care nu sunt caracteristice bazinului hidrografic respectiv. Luand in consideratie acest fapt, se impune evaluarea distributiei si caracterizarea variabilitatii genetice a salmonidelor din Romania. Pana in prezent, in Romania, nu s-a efectuat un studiu unitar al salmonidelor din punct de vedere genetic, studiu absolut necesar pentru identificarea speciilor autohtone, alohtone sau hibride.
STABILIREA BAZINELOR HIDROGRAFICE CU PREZENTA PERMANENTA SAU SPORADICA A SALMONIDELOR SALBATICE
Pastravul indigen (comun) - Salmo trutta fario L.
Este specia nativa cu cea mai mare raspandire in Europa. A fost introdus si in India, Australia, America de Nord si de Sud, Madagascar. Pastravul comun este un peste foarte polimorfic, prezentand mai multe "subspecii", care reprezinta de fapt populatii cu distributie geografica si comportament distinct.
Este un salmonid stenotermal, care prefera apele limpezi, reci (12-16 sC) si puternic oxigenate. Nu suporta mari variatii de temperatura. Reproducerea are loc la sfarsitul toamnei sau inceputul iernii cand migreaza in amonte si isi depune icrele pe un teren cu pietris fin. Puietul eclozeaza in perioada 15 martie - 15 aprilie.
In Romania este intalnit in mai toate cursurile de apa din zona montana, populeaza lacurile naturale si artificiale, inclusiv unele lacuri alpine din Muntii Fagaras, Parang si Retezat. Dupa adaptarea la cresterea in sistem intensiv a fost realizat cel mai amplu program de populare si repopulare a apelor de munte cu aceasta specie. Habitatele acestei specii sunt prezentate in hartile din Anexa I.
Pastravul fantanel - Salvelinus fontinalis M.
Pastravul fantanel este originar din zona de coasta atlantica a Americi de Nord. A fost introdus in Europa, India, America de Sud, Australia, Japonia, Rusia, etc. In Romania a fost introdus initial in Moldova in jurul anului 1906 (Decei, 2001) si a populat apoi si cateva rauri din Transilvania. La momentul actual efectivele din liber sunt foarte reduse, specia fiind crescuta in pastravarii pentru consum.
Reproducerea are loc in general in lunile octombrie si noiembrie. Femelele pot produce intre 2500 - 5000 icre/kg corp, in functie de marimea si varsta fiecareia. In functie de temperatura apei, ecloziunea puietului are loc dupa 2-3 luni. Pastravul fantanel atinge maturitatea la 2-3 ani si media de viata este de 6 ani. Zonele populate cu pastrav fantanel sunt prezentate in Anexa I.
Pastravul de mare - Salmo trutta labrax P.
Reprezinta o specie marina, endemica bazinului Pontic, intalnita in Marea Neagra si Azov. Prefera zona de coasta si riurile mari limitrofe. Specia a fost semnalata in Caucaz, Crimea, Asia Mica, sudul Ucrainei, Turcia, Bulgaria. In Romania au fost inregistrate aparitii sporadice in zona litoralului romanesc si pe Dunare, fara sa urce prea mult in amonte. Actualmente specia se captureaza intr-un numar mic de exemplare pe litoralul Marii Negre, de la Sfantu Gheorghe spre sud si sporadic in Dunare si in zona Razim-Sinoie.
Atinge varsta maturitatii sexuale la 3-4 ani. In cazul speciei Salmo trutta labrax se poate vorbi despre o migratie de reproducere din Marea Neagra in fluvii care are loc primavara (la sfarsitul lunii aprilie) cu o posibila intoarcere in mare dupa depunerea icrelor si o deplasare a subadultilor din mediul marin in cel dulcicol.
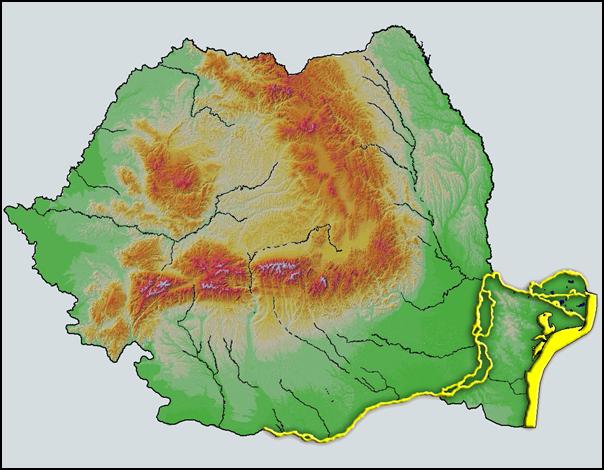
Figura 2. Harta raspandirii actuale a pastravului de mare (Salmo trutta labrax, Pallas 1820) pe teritoriul Romaniei.
Inca de la semnalare, pastravul de Marea Neagra a fost rar si nu a reprezentat niciodata vreo importanta economica pentru pescuitul romanesc. Literatura de specialitate semnaleaza aceasta specie la noi in tara in Marea Neagra (habitat de hranire si crestere), pe bratele si in lacurile din delta si pe Dunare pana in dreptul Km 700 (Banarescu 1964, Svetovidov 1964, Otel 2007).
Alaturi de lostrita, pastravul de Marea Neagra (Salmo trutta labrax, Pallas 1811), sub-endemit pontic inclus in Lista Rosie a Rezervatiei Biosferei Delta Dunarii (Otel 2000; Otel 2007), este o specie periclitata de salmonide din tara noastra cu potential real pentru dezvoltarea salmoniculturii, care necesita evaluarea starii stocului si adoptarea de masuri urgente de conservare.
In figura 2 prezentam harta raspandirii actuale a pastravului de Marea Neagra pe teritoriul Romaniei. Harta a fost realizata coroborand informatiile din toate publicatiile existente (Antipa, 1909; Banarescu, 1964; Carausu, 1962; Kottelat, 2007; Otel, 2000; Popescu-Gorj, 1956; Radu, S. 2008 a,b), observatiile proprii de la coasta marii Negre si complexul Razim - Sinoie, ca si semnalari si observatii ale unor pescari profesionisti si pescari sportivi.
Pastravul curcubeu - Oncorhynchus mykiss M.
Pastravul curcubeu isi are originea in zona nord-estica a Pacificului. Datorita adaptabilitatii, a fost introdus in Europa, Asia, America de Sud, Africa, etc. Specia se preteaza cel mai bine pentru cresterea in sistem intensiv pentru consum.
In ceea ce priveste reproducerea artificiala, in decursul anilor aceasta specie a fost incrucisata selectiv rezultand un numar destul de mare de varietati de O. mykiss, diferenta principala intre acestea fiind sezonul de depunere a icrelor. Majoritatea se reproduc in perioada ianuarie - mai, dar exista cazuri in care reproducerea are loc in decembrie sau chiar mai devreme. In Romania a fost introdus in Moldova, Maramures, Banat si Transilvania in jurul anului 1965 (Decei, 2001). In prezent efectivele din liber sunt foarte reduse, ajungand in rau accidental, din pastravarii. In Anexa I sunt prezentate habitatele populate cu pastrav curcubeu din zona Munteniei si hartile cu distributia geografica a acestei specii.
Lipanul - Thymallus thymallus L.
Este originar din Europa, iar in Romania specia este bine reprezentata in apele noastre. Este singurul salmonid autohton nealterat din punct de vedere genetic deoarece nu s-au facut importuri de material biologic pentru aceasta specie.
Ocupa partea inferioara a apelor de munte, unde apele sunt mai adanci, mai linistite si cu albia pietroasa. Populatiile de lipan din tara noastra sunt impartite in zona de nord-est (Neamt-Suceava), zona de curbura (Buzau-Covasna), sud (Arges-Prahova) si vest (Mures). Distributia geografica a lipanului din zona Munteniei este prezentata in Anexa I.
Lostrita - Hucho Hucho L.
Este o specie endemica bazinului dunarean care se intalneste majoritar in regiunea superioara a Dunarii, dar si in majoritatea afluentilor sai mari. Lostrita traieste in ape curgatoare repezi si migreaza in amonte pe distante mici in sezonul de imperechere. Poate depasi la maturitate lungimea de 1 m.
La
noi lostrita era raspandita in trecut in Dunare, Olt,
Mures, Lotru, Jiu, Arges si Cerna si foarte probabil in
Crisul Repede, Buzau, Moldova si Timis. Azi se mai
gaseste in raul Bistrita, de
ANALIZE FIZICO-CHIMICE PRIVIND CALITATEA APEI HABITATELOR STUDIATE
In prima parte a fost realizata o trecere in revista a situatiei calitatii apei Dunarii, principalul habitat de reproducere si crestere a puilor si juvenililor de pastrav de Marea Neagra, pentru perioada 2005 - 2009.
Dinamica multianuala a valorilor de pH pe Dunare si brate
Domeniul optim de pH este cuprins intre 6,5 si 8,5. Valorile medii anuale de pH se incadreaza in acest domeniu, exceptie facand Cotul Pisicii in anul 2009 si Aval Izmail in anul 2007.
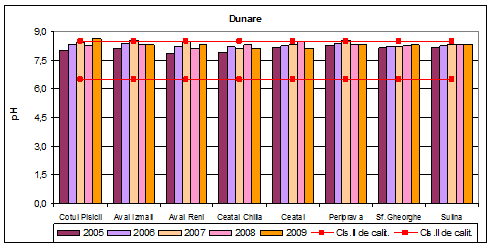
Figura 3. Dinamica multianuala a valorilor de pH pe Dunare si brate.
Dinamica multianuala a oxigenului dizolvat pe Dunare si brate
Concentratiile medii anuale determinate pentru oxigenul dizolvat, in dinamica multianuala, din probele de Dunare, au valori corespunzatoare clasei a II-a de calitate (stare ecologica buna) atat in anul 2009 cat si in anii anteriori.
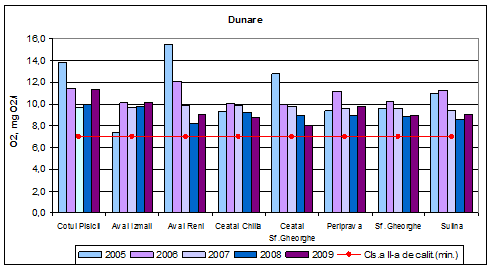
Figura 4. Dinamica multianuala a oxigenului dizolvat pe Dunare si brate.
Dinamica multianuala a consumului biochimic de oxigen pe Dunare si brate
Concentratiile medii anuale ale consumului biochimic de oxigen la 5 zile are valori ce incadreaza apa de suprafata din Dunare, in anul 2009 in clasa a II-a de calitate, exceptie facand valorile determinate la Cotul Pisicii si Aval Izmail, unde s-au determinat valori corespunzatoare clasei a III-a de calitate.
In anii 2006, 2007 si 2008 Dunarea si bratele acesteia se incadreaza in clasa a II-a de calitate, cu mici exceptii si anume Cotul Pisicii (2006 -2007) si Aval Reni (2007), cand s-au obtinut valori corespunzatoare clasei a III-a de calitate.
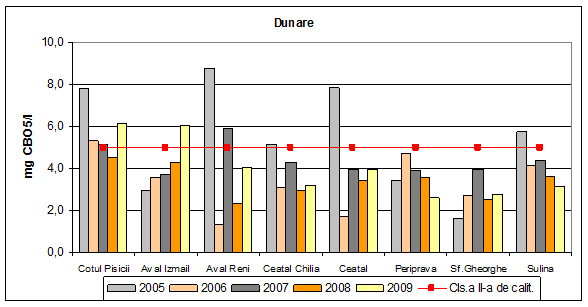
Figura 5. Dinamica multianuala a consumului biochimic de oxigen pe Dunare si brate.
Dinamica multianuala a indicelui de permanganat pe Dunare si brate
In anul 2009, valorile concentratiilor indicelui de permanganat exprimat in mg O2/l, incadreaza apa Dunarii si bratelor acesteia in clasa I de calitate. In anii anteriori, valorile indicelui de permanganat incadreaza apa Dunarii si bratelor acesteia in clasa I de calitate, exceptie facand Cotul Pisicii in 2008 (clasa a II-a de calitate) si 2006 (clasa a III-a de calitate), in clasa a II-a de calitate incadrandu-se de asemenea in 2005, Ceatal Sf. Gheorghe, in 2006 Cotul Pisicii si Sf. Gheorghe, iar in 2008 Aval Izmail, Ceatal Chilia, Ceatal Sf. Gheorghe, Periprava si Sulina.
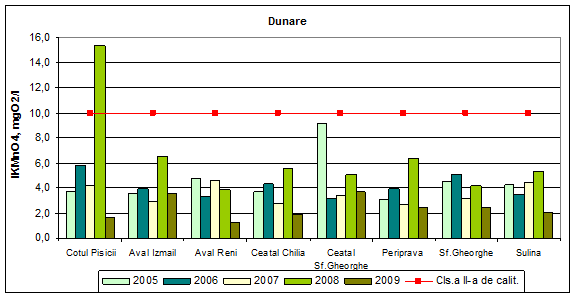
Figura 6. Dinamica multianuala a indicelui de permanganat pe Dunare si brate.
Dinamica multianuala a azotului amoniacal pe Dunare si brate
In anul 2009, valorile concentratiilor de azot amoniacal determinate din punctele de prelevare selectate, au valori corespunzatoare clasei I de calitate, respectiv stare ecologica foarte buna. Aceeasi dinamica este observata si in anii 2006, 2007 si 2008 cu o singura exceptie si anume Aval Reni in anul 2006. In anul 2005 valorile determinate pentru azot amoniacal sunt valori corespunzatoare claselor I si a II-a de calitate.
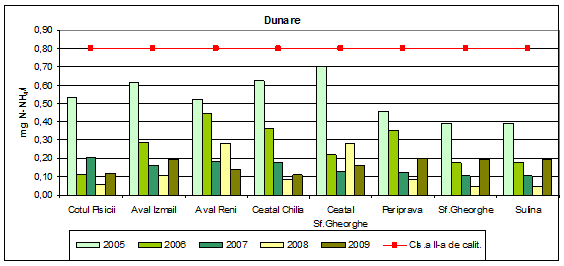
Figura 7. Dinamica multianuala a azotului amoniacal pe Dunare si brate.
Dinamica multianuala a azotului din azotiti pe Dunare si brate
Valorile medii determinate pentru azotul din azotiti in anul 2009 incadreaza apa din Dunare si brate in clasa a II-a de calitate, exceptie facand Periprava si Sf. Gheorghe (clasa a III-a de calitate) si Sulina (clasa a IV-a de calitate). In anii 2006, 2007 si 2008 valorile determinate sunt valori corespunzatoare clasei a II-a de calitate, (stare ecologica buna), exceptie facand in 2006 Aval Reni si Sf. Gheorghe. Valorile determinate in anul 2005 sunt valori corespunzatoare clasei a IV-a si respectiv a V-a de calitate.
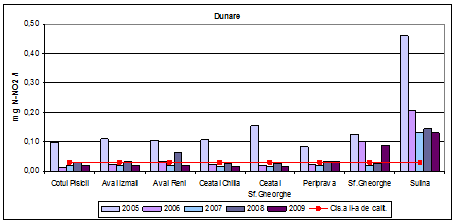
Figura 8. Dinamica multianuala a azotului din azotiti pe Dunare si brate.
Dinamica multianuala a azotului din azotati pe Dunare si brate
In anul 2009, valorile medii ale concentratiilor de azot din azotati sunt corespunzatoare claselor a III-a, a IV-a si chiar a V-a de calitate.
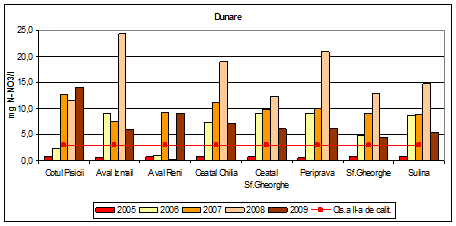
Figura 9. Dinamica multianuala a azotului din azotati pe Dunare si brate.
Comparand anii 2005, 2006, 2007 si 2008 se observa o crestere a concentratiilor de azot din azotati, in anul 2008 obtinandu-se valori corespunzatoare clasei a V-a de calitate (stare ecologica foarte proasta).
Dinamica multianuala a azotului total pe Dunare si brate
Concentratia medie anuala a azotului total determinat din probele de apa in anul 2009, incadreaza apa Dunarii si a bratelor acesteia in clasele a III-a, a IV-a si a V-a de calitate (stare ecologica moderata, proasta si foarte proasta). Valorile concentratiilor de azot total determinate, in anul 2008, incadreaza apa Dunarii si a bratelor acesteia in clasa a V-a de calitate, exceptie facand Aval Reni (clasa a II-a de calitate) si Cotul Pisicii (clasa a IV-a de calitate). Comparativ, in anul 2007, apele sunt incadrate in clasa a III-a de calitate, exceptie facand Cotul Pisicii si Periprava (clasa V de calitate) si Ceatal Chilia (clasa IV de calitate).
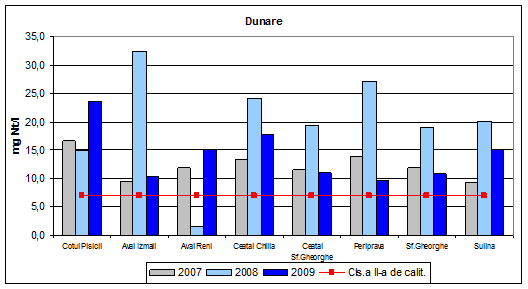
Figura 10. Dinamica multianuala a azotului total pe Dunare si brate.
Dinamica multianuala a ortofosfatilor solubili pe Dunare si brate
Valoarea limita stabilita pentru clasa a II-a de calitate (stare ecologica buna) pentru ortofosfati solubili exprimati ca P-PO4-3 este de 0,2 mg P/L. Valorile concentratiilor medii anuale obtinute in apa Dunarii si a bratelor ei, incadreaza aceste ape in perioada 2005-2009, in clasa I de calitate.
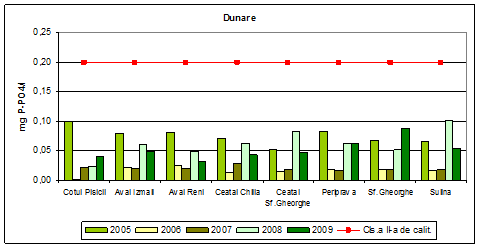
Figura 11. Dinamica multianuala a ortofosfatilor solubili pe Dunare si brate.
Dinamica multianuala a fosforului total pe Dunare si brate
In anul 2009, valorile concentratiilor de fosfor total incadreaza apa Dunarii si a bratelor acesteia in clasa I de calitate. Aceeasi incadrare s-a obtinut si in anii 2006, 2007 si 2008. Valorile concentratiilor de fosfor total in anul 2005, sunt valori corespunzatoare clasei III de calitate (exceptie Ceatal Chilia, Ceatal Sf. Gheorghe, Cotul Pisicii- clasa II de calitate).
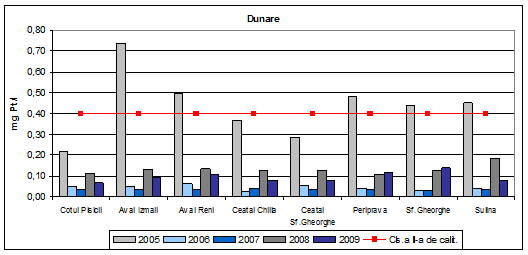
Figura 11. Dinamica multianuala a fosforului total pe Dunare si brate.
Dinamica multianuala a sulfatilor pe Dunare si brate
In anul 2009 valorile medii determinate pentru sulfati incadreaza apa din Dunare si brate in clasa I de calitate. Concentratiile medii anuale determinate pentru sulfati in anii 2005, 2007 si 2008 incadreaza apa din Dunare si brate in clasa I de calitate (stare ecologica foarte buna), exceptie facand Cotul Pisicii, Aval Izmail, Periprava si Sf. Gheorghe in anul 2005 si Ceatal Sf. Gheorghe si Sulina in anul 2008, cand s-au inregistrat valori ale concentratiilor de sulfati mai mari de 60 mg/L, ceea ce incadreaza aceasta apa in clasa a II-a de calitate, respectiv stare ecologica buna.
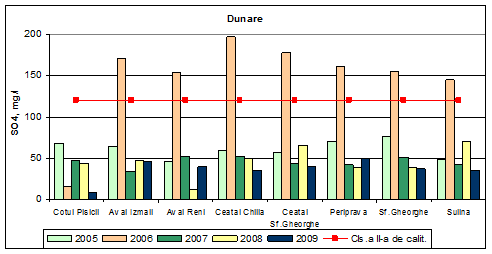
Figura 13. Dinamica multianuala a sulfatilor pe Dunare si brate.
Dinamica multianuala a concentratiei de crom pe Dunare si brate
Valorile medii ale concentratiei de crom incadreaza apa Dunarii in anul 2009 in clasa I de calitate, exceptie facand Ceatal Sf. Gheorghe (clasa a II-a de calitate) si Sulina (clasa a III-a de calitate).
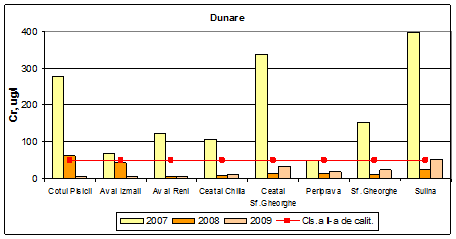
Figura 14. Dinamica multianuala a concentratiei de crom pe Dunare si brate.
In anul 2008, valorile concentratiilor de crom obtinute incadreaza apa Dunarii in clasa I de calitate cu exceptia punctelor Cotul Pisicii (clasa a III-a) si Aval Izmail (clasa a II-a de calitate). In anul 2007, in clasa a II-a de calitate se incadreaza Periprava, in clasa a III-a de calitate se incadreaza Aval Izmail, in clasa a IV-a de calitate se incadreaza Aval Reni, Ceatal Chilia si Sf.Gheorghe, iar in clasa a V-a se incadreaza Ceatal Sf.Gheorghe si Sulina.
Dinamica multianuala a concentratiei de cupru pe Dunare si brate
In anul 2009, concentratiile de cupru determinate in apa Dunarii si a bratelor, incadreaza apa in clasele I de calitate (Cotul Pisicii, Ceatal Sf. Gheorghe si Sf. Gheorghe), a II-a de calitate (Ceatal Chilia si Sulina), a III-a de calitate (Aval Izmail) si a IV-a de calitate (Periprava). In functie de mediile anuale de cupru, in anul 2008, in clasa I de calitate se incadreaza Aval Izmail, Ceatal Chilia, Cotul Pisicii si Sulina, in clasa a II-a de calitate se incadreaza Aval Reni, Ceatal Sf.Gheorghe, si Sf. Gheorghe, iar in clasa a V-a de calitate se incadreaza Periprava. In anul 2007, apele se incadreaza in clasa a IV-a de calitate, exceptie facand Ceatal Sf.Gheorghe (clasa a V-a de calitate). In anul 2006, concentratiile de cupru determinate in apa Dunarii si a bratelor, incadreaza apa in clasa a V-a de calitate.
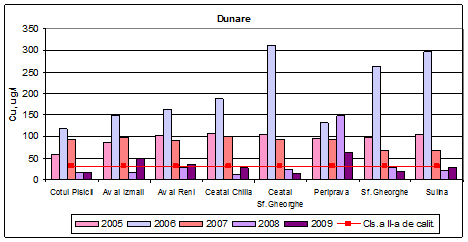
Figura 15: Dinamica multianuala a concentratiei de cupru pe Dunare si brate.
Dinamica multianuala a concentratiei de zinc pe Dunare si brate
Pe toata perioada studiata, 2005-2009, valorile concentratiilor de zinc din apa Dunarii si a bratelor, incadreaza aceste ape in clasa I de calitate, cu o singura exceptie si anume in anul 2006, Aval Reni (clasa a II-a de calitate).

Figura 16: Dinamica multianuala a concentratiei de zinc pe Dunare si brate.
Dinamica multianuala a concentratiei de arsen pe Dunare si brate
Valorile concentratiilor de arsen din apa Dunarii si a bratelor, incadreaza aceste ape in clasa a II-a de calitate, cu doua exceptii si anume Ceatal Chilia si Periprava, ale caror valori de concentratie incadreaza aceste ape in clasa a III-a de calitate. Arsenul determinat in anul 2008, incadreaza apa bratelor in clasa a II-a de calitate, exceptie facand Sf. Gheorghe (clasa a III-a de calitate). In anul 2007 apa bratelor se incadreaza in clasa I de calitate, exceptie facand Aval Reni (clasa a II-a de calitate).
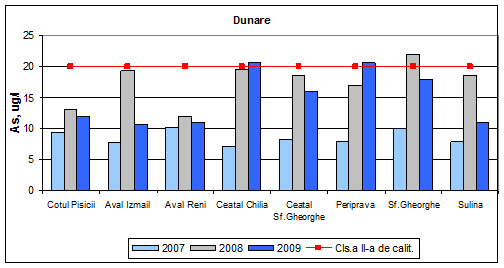
Figura 17. Dinamica multianuala a concentratiei de arsen pe Dunare si brate.
Dinamica multianuala a concentratiei de plumb pe Dunare si brate
Valorile mediilor anuale ale concentratiilor de plumb incadreaza apa Dunarii in anul 2009 in toate cele cinci clase de calitate. Aceeasi incadrare a acestor ape se observa si pentru perioada 2005-2008. In anul 2008, valorile mediilor anuale ale concentratiilor de plumb incadreaza apa Dunarii in clasa I de calitate, exceptie facand Periprava (clasa a IV-a de calitate) si Sulina (clasa a II-a de calitate). In anii 2006 si 2007, valorile concentratiilor medii anuale incadreaza apa in clasa a V-a de calitate, exceptie facand Aval Reni (2006) si Sf. Gheorghe (2007) ale caror valori de concentratie incadreaza apa in clasa a IV-a de calitate. In anul 2005 se inregistreaza o variatie a concentratiilor medii anuale ce incadreaza apa in clasele a III-a, a IV-a si a V-a de calitate.
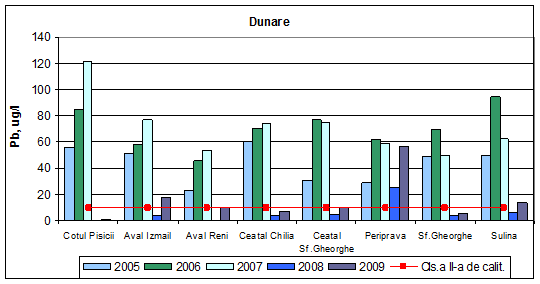
Figura 18. Dinamica multianuala a concentratiei de plumb pe Dunare si brate.
Dinamica multianuala a concentratiei de cadmiu pe Dunare si brate
Valorile concentratiilor de cadmiu incadreaza apa Dunarii si a bratelor in clasa a V-a de calitate, respectiv stare ecologica foarte proasta. Aceeasi incadrare o gasim si in anii 2005-2008 exceptie facand Periprava (2007), Sulina (2005), Sf. Gheorghe (2005), Cotul Pisicii (2005), Ceatal Chilia (2005), Aval Reni (2005), cand au fost determinate valori ale concentratiilor anuale de cadmiu ce incadreaza apa in clasa a IV-a de calitate (stare ecologica proasta). O stare ecologica buna (clasa a II-a de calitate) s-a determinat in anul 2006 la Cotul Pisicii. In punctul Aval Reni (2006), s-au determinat valori medii anuale ale concentratiilor de cadmiu ce incadreaza apa in clasa a III-a de calitate (stare ecologica moderata).
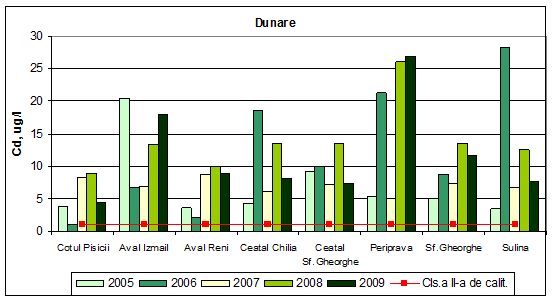
Figura 19. Dinamica multianuala a concentratiei de cadmiu pe Dunare si brate.
Dinamica multianuala a concentratiei de fier pe Dunare si brate
Valorile concentratiilor de fier din apa Dunarii si a bratelor, in anul 2009, incadreaza apa acestora in clasele a II-a (Ceatal Chilia, Periprava si Sf. Gheorghe), a III-a (Cotul Pisicii, Aval Reni si Sulina) si a IV-a (Aval Izmail si Ceatal Sf. Gheorghe) de calitate.
De asemenea, in anul 2008 se inregistreaza o variatie a valorilor medii anuale ale concentratiilor de fier ce incadreaza apa bratelor Dunarii in toate cele cinci clase de calitate: clasa I (Periprava), clasa a II-a (Aval Reni), clasa a III-a (Cotul Pisicii), clasa a IV-a (Ceatal Chilia, Ceatal Sf. Gheorghe, Sf. Gheorghe si Sulina) si clasa a V-a (Aval Izmail). In anul 2007 s-au determinat valori ale concentratiilor de fier ce incadreaza apa Dunarii in clasa a III-a de calitate (stare ecologica moderata), cu o singura exceptie si anume Ceatal Chilia unde s-au inregistrat valori ale concentratiilor medii anuale de fier ce incadreaza apa in clasa I de calitate (stare ecologica foarte buna). In anii 2005-2006, valorile mediilor anuale ale concentratiilor de fier incadreaza apa bratelor Dunarii in clasele a IV-a si a V-a de calitate adica o stare ecologica proasta si foarte proasta.
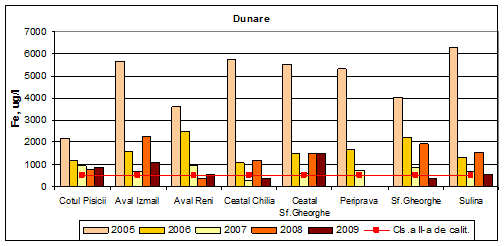
Figura 20. Dinamica multianuala a concentratiei de fier pe Dunare si brate.
Dinamica multianuala a concentratiei de mangan pe Dunare si brate
In anul 2009, valorile concentratiilor de mangan din apa Dunarii si a bratelor, incadreaza aceste ape in clasa I de calitate, respectiv stare ecologica foarte buna, cu mici exceptii si anume, clasa a II-a de calitate (Sulina) si clasa a III-a de calitate (Aval Izmail). Valorile mediilor anuale ale concentratiilor de mangan incadreaza apa bratelor Dunarii in anul 2008 in clasa a III-a de calitate, cu exceptia punctelor Periprava, Cotul Pisicii si Aval Reni, unde concentratiile de mangan au valori medii anuale corespunzatoare clasei I de calitate (stare ecologica foarte buna). In anii 2005-2007 s-au determinat valori medii anuale ale concentratiilor de mangan ce incadreaza apa in clasele a III-a (stare ecologica moderata) si a IV-a de calitate (stare ecologica proasta).
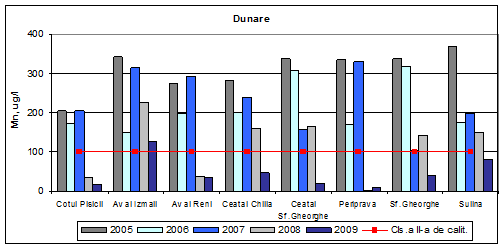
Figura 21. Dinamica multianuala a concentratiei de mangan pe Dunare si brate.
Dinamica multianuala a concentratiei de nichel pe Dunare si brate
Valorile concentratiilor de nichel din apa Dunarii si a bratelor in anul 2009, incadreaza aceste ape in clasele a II-a, a III-a si chiar a IV-a de calitate, respectiv stare ecologica buna, moderata si chiar proasta.
In anul 2008 se inregistreaza o variatie a valorilor mediilor anuale ale concentratiilor de nichel ce incadreaza apa bratelor Dunarii in patru din cele cinci clase de calitate: clasa I (Aval Reni), clasa a II-a (Sf. Gheorghe, Periprava si Ceatal Chilia), clasa a III-a (Sulina, Ceatal Sf. Gheorghe si Aval Izmail) si clasa a V-a (Cotul Pisicii). In anul 2007, starea ecologica a apei bratelor Dunarii este corespunzatoare claselor a III-a, a IV-a si a V-a de calitate.
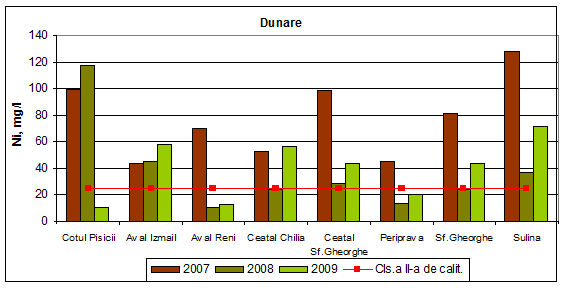
Figura 22. Dinamica multianuala a concentratiei de nichel pe Dunare si brate.
Dinamica multianuala a concentratiei de mercur din Dunare si brate
Valorile concentratiilor de mercur determinate in anul 2009 in apa Dunarii si a bratelor, incadreaza aceste ape in clasa I de calitate.
In anul 2008, mercurul determinat din apa bratelor Dunarii, are valori corespunzatoare clasei I de calitate. Aceeasi dinamica se observa si in anii 2006 si 2007, exceptie facand Sf. Gheorghe in 2006 (clasa a II-a de calitate).
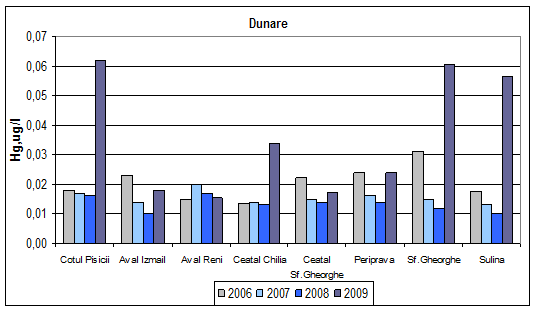
Figura 23. Dinamica multianuala a concentratiei de mercur pe Dunare si brate.
Dinamica multianuala a concentratiei de lindan (gamma HCH) pe Dunare si brate
In Normativul 161/2006 stabileste standarde de calitate pentru substantele periculoase si prioritar periculoase. Astfel pentru lindan, standardul de calitate este de 0,02 µg/L, raportandu-ne la aceasta valoare, observam ca nu sunt depasiri in perioada 2005-2008.
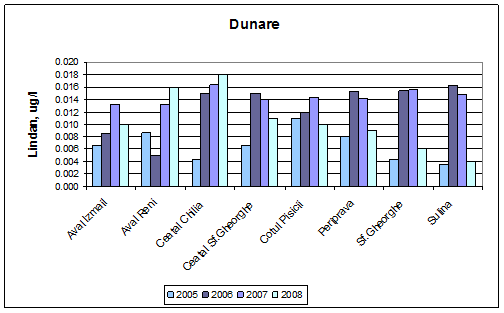
Figura 24. Dinamica multianuala a concentratiei de lindan (gamma HCH) pe Dunare si brate.
Dinamica multianuala a concentratiei de pp' DDT pe Dunare si brate
Standardul de calitate pentru pp' DDT este 0,01 µg/L. Aceasta valoare nu este depasita in perioada 2005-2008 in apa bratelor Dunarii.
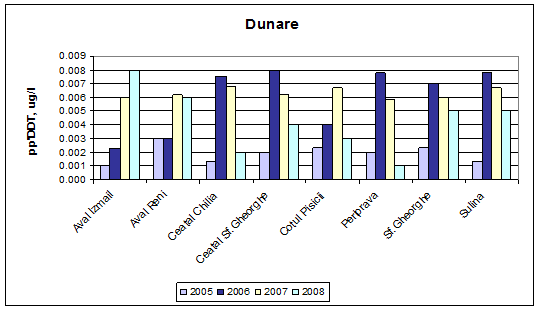
Figura 25. Dinamica multianuala a concentratiei de pp' DDT pe Dunare si brate.
Dinamica multianuala a concentratiei de PCB-uri
In perioada 2007-2009, s-au facut determinari de PCB-uri (compusi bifenilici policlorurati), respectiv PCB 28, 52, 101, 118, 138, 153, 180. Valorile concentratiilor determinate sunt sub limita de detectie a aparatului.
Din aceasta trecere in revista (2005 - 2009) se constata deficiente grave ale calitatii apei Dunarii in privinta continutului de:
azotiti la statiile situate aproape de varsarea in Marea Neagra,
azot din azotati,
- azot total,
- crom, in anul 2007,
- cupru, in anul 2007,
- plumb, cadmiu, fier si mangan, elemente la care starea ecologica a apei Dunarii in perioada analizata se incadreaza in categoria proasta si foarte proasta.
Distributia salmonidelor pe bazine hidrografice si analizele fizico-chimice ale apelor
In partea a doua a realizat un studiu al habitatelor populate cu celelalte specii de salmonide, evaluandu-se proprietatile fizico-chimice ale apelor si identificandu-se factorii limitativi ai acestor areale.
Identificarea factorilor limitativi ai habitatelor populate cu salmonide
1. Factori limitativi antropici
Barajele amenajate - sunt constructiile care au fragmentat cursurile de apa si reprezinta cauza principala a declinului populatiilor de salmonide, aceste specii de pesti avand un puternic caracter migrator, in perioada de "boiste" exemplarele mature urcand pe rau catre izvoare si afluentii principali pentru a se reproduce.
Datorita expansiunii demografice si regularizarii cursului principal, constructiile au coborat mult spre albia minora a raurilor, pagubele produse la inundatii sau viituri impunand si regularizarea unor afluenti prin lucrari de corectare a torentilor. Astfel au fost distruse putinele locuri de reproducere ramase disponibile pentru salmonide.
Majoritatea constructiilor hidrotehnice nu au instalatii pentru migratia si circulatia pestelui, cunoscute sub denumirea de "scari pentru pesti". Acolo unde exista, in mare majoritate sunt prost concepute, neintretinute sau scoase din functiune.
Intotdeauna prin constructia unui baraj, in special cel cu rol hidroenergetic, intreg habitatul din aval se modifica radical. In timp ce materia organica este depozitata in lac si incepe eutrofizarea acestuia, in aval lantul trofic este intrerupt, dereglarile populationale pot duce la disparitia unor specii, se creaza conditii si nise pentru alte specii care pot prolifera si elimina speciile nepregatite sau care nu se pot apara in fata acestora.
Datorita conditiilor climatice din ultima perioada, debitele raurilor s-au schimbat semnificativ, o mare parte din acumularile existente au utilizare complexa, atat energetica dar si ca sursa de apa potabila, desi eutrofizarea si incarcatura microbiologica care se produce in timp poate impune renuntarea la aceasta a doua utilizare.
Aceasta utilizare genereaza in aval de baraj debite semnificativ reduse si cu fluctuatii frecvente care pun in pericol populatiile salmonicole astfel, se restrange habitatul si se formeaza concentratii de efective pe anumite zone s-au chiar punctiform unde sunt decimate usor de pradatori sau de pescuitul irational.
Sunt tari in Europa unde amenajarile hidrotehnice pentru ameliorarea torentilor si care intrerup habitatele acvatice sunt demolate, cautandu-se aducerea cursului de apa la conditiile naturale. Se cauta solutii pentru drenarea materiei care produce colmatarea lacurilor artificiale si introducerea acesteia in circuitul din aval al raului.
Pragurile artificiale sunt construite cu scopul de a reduce eroziunea apei datorata vitezei mari de scurgere si pentru a restrange latimea unui rau. Aceste amenajari au fost colmatate sau dislocate obligand raul la un alt traseu, iar prin inaltimea si amenajarea la distante necorespunzatoare au devenit obstacole in calea pestilor. Actiunile de amenajare a raurilor au generat o paradigma «omul contra naturii», deciziile au fost mai mult ingineresti fortand raul sa devina un element static, iar in final pierderile au depasit cu mult beneficiile. Consideram ca este esential ca pe viitor, la intocmirea schemelor cadru de amenajare si gospodarire a apelor, sa participe si specialisti din domeniul piscicol.
Exploatarea pietrisului si nisipului din albia raului - reprezinta o forma de «mutilare» a unui curs de apa, distrugandu-se astfel locurile de adapost ale pestelui adult si astfel puietul nu mai beneficiaza de zone lentice. Zonele malurilor, unde se dezvolta vegetatia acvatica, cat si de mal, sunt distruse, nevertebratele care constituie hrana principala a pestelui din perioada larvara, nu mai au conditii de dezvoltare, si astfel apare mortalitatea in randul puietilor, modificandu-se structura de varsta a populatiilor. Totodata, ca orice activitate care implica utilaje, oameni si intrarea in apa, devine o sursa potentiala de poluare accidentala.
Exploatarile forestiere - in ultima perioada, datorita lacunelor legislative, exploatarile de masa lemnoasa de catre agenti economici fara nicio pregatire in domeniu, taierile abuzive si pe suprafete mari, nerespectarea regulamentelor si clauzelor contracuale privind taierea si colectarea materialului lemnos, au produs efecte de domino asupra habitatelor. Despaduririle masive au condus la degradarea solului, formarea de viituri, inundasii si modificari semnificative ale cursurilor de apa. Este cunoscut faptul ca o perioada indelungata cu turbiditate ridicata a apei poate provoca mortalitate prin asfixiere, crearea unor viituri puternice in care este angrenat si material solid (bolovani, lemne, deseuri) si impingerea populatiei piscicole in aval, obligand-o sa paraseasca locurile favorabile. Refacerea efectivelor si habitatului necesita ani buni si o protectie constanta.
Prelucrarea lemnului - in special debitatul cherestelei, o activitate strans legata de cea prezentata anterior, rumegusul aruncat in rauri sau depozitat pe maluri, are efecte negative asupra pestilor.
Rumegusul se fixeaza intre lamelele branhiale ale pestelui, iar in cantitati mari produce asfixieri sau leziuni majore care devin porti de intrare pentru bacterioze sau alti paraziti. Puietul este afectat si indirect, prezenta rumegusului in cantitati mari sau spalarea lui din zona zona malurilor, unde este depozitat, de catre ploi, duce la cresterea aciditatii pe zona respectiva, hrana puietului se reduce sau chiar dispare, efectivele salmonicole avand de suferit inca din aceasta faza de dezvoltare.
Daca in ultimul timp costurile pentru combustibilii fosili au fortat utilizarea rumegusului la incalzirea locuintelor, ramane in continuare problema gaterele ilegale amplasate in zona superioara a cursurilor de apa.
Poluarea chimica - este frecventa si poate fi accidentala sau discontinua, in special cu ape uzate care pot genera unde soc de poluare cu diversi compusi chimici. Poluarea cu substante chimice este devastatoare pentru populatiile piscicole si nu numai, activitatile industriale fiind principala sursa generatoare.
O sursa majora de poluare o reprezinta haldele de steril rezultat in urma exploatarilor miniere, situatia fiind critica in Maramures-Baia Borsa unde exploatarile si industria prelucratoare de metale neferoase au generat poluari cu metale grele si dupa inchiderea exploatarilor, iar rezidurile cianurice ajunse in rauri au creat probleme transfrontaliere. Este cunoscut faptul ca o cantitate de zinc mult peste limita admisibila si pe o perioada indelungata afecteaza gonadele pestilor si implicit reproducerea.
Poluarea cu compusi organici biodegradabili este cea mai frecventa prin apele menajer-fecaloide rezultate din activitatile casnice si industriale (producerea celulozei, industria alimentara, etc.). Daca la acesta situatie se adauga si poluarea cu substante toxice, degradarea substantelor organice este redusa semnificativ deoarece flora microbiologica care realizeaza biodegradarea acestora este inhibaa sau distrusa.
In zonele rurale, foarte frecvent, gunoiul menajer este aruncat sau depozitat in apropierea malurilor, astfel diverse substante chimice ajung in rau.
Ploile acide - principala sursa este arderea combustibililor fosili si degajarea in atmosfera a bioxidului de sulf si oxizilor de azot, ulterior, prin procese complexe ce au loc la nivel atmosferic, se genereaza acidul sulfuric si azotic sau aerosoli de azotat si sulfat, acestia ajungand in apa si pe sol prin precipitatii, ceata, chiciura sau particule de aerosoli.
Termenul de ploaie acida a fost introdus de chimistul Robert Angus Smith in anul 1872, observand cum ploaia afecteaza plantele si cladirile. Abia in anii '50 s-a constatat ca problema este transfrontaliera.
O alta sursa care genereaza aciditate, este poluarea cu amoniu (accidental sau din activitatea industriala) pe care o serie de bactrerii il transforma rezultand ioni de hidrogen, acest fapt marind aciditatea apei. Exploatatiile miniere, in special pirita expusa la aer si umiditate, elibereaza mari cantitati de H+ care conduc in final la marirea aciditatii apelor.
Acidifierea apelor nu apare sau este puternic redusa in zonele calcaroase, neutralizarea aciditatii fiind realizata de prezenta calciului si magneziului.
Braconajul si pescuitul excesiv - reprezinta factori de presiune asupra populatiilor de salmonide. Braconajul piscicol imbraca diverse forme avand intensitati difetite. Folosirea de scule si unelte interzise cum ar fi ostia, priponul, postrovolul, plase si setci monofilament, utilizarea substantelor otravitoare sau toxice, folosirea curentului electric, explozivilor, abaterea cursului de apa, constitue moduri si tehnici frecvente utilizate de braconieri.
Se practica des in apele de munte si in special la lostrita, braconajul cu lumina si ostia in perioada de boiste. Exemplarele sunt usor de capturat pentru ca acestea urca in zone cu apa mai mica unde devin usor de prins. Locurile de pescuit sunt pastrate de generatii si sunt cunoscute de localnici sau pescarii impatimiti. Deoarece se scot exemplarele cele mai mari, adulte, pierderile sunt imense, avand in vedere ca o femela de 5 kg depune peste 10000 de icre, pierzandu-se astfel generatii intregi.
Pescuitul excesiv, fara a tine seama de perioada admisa, dimensiunile pestelui, numarul de exemplare pastrate, folosirea momelilor interzise, contribuie si ele la reducerea efectivelor. Tot unor pescari datoram si popularea unor lacuri montane cu specii de ciprinide si nu numai, acestia folosind ca momeala peste viu in capturarea salmonidelor. Dupa terminarea partidei de pescuit pestele viu ramas a fost aruncat in lac fara sa se gandeasca la consecinte.
2. Factori limitativi naturali
Vidra (Lutra lutra L.) este pradatorul natural principal pentru salmonidele din liber dar si din ferme. Traieste in apropierea apei, consuma numai peste (1 kg/zi) hranindu-se mai mult noaptea. Daca cursul de apa nu mai ofera hrana vidra se muta in alta zona, uneori face adevarate expeditii terestre pana la o alta zona.
Nurca (Lutreola lutreola L.) seamana cu vidra dar are talie mult mai redusa si produce pagube insemnate, se instaleaza in special in zona fermelor.
Soarecele cu bot ascutit (Neomis fodiens Pall) denumit si chitcanul de apa, este de culoare negru-brun, alb murdar pe piept si are botul lunguiet si ascutit. Traieste in malul apelor, consuma cu predilectie puiet si produce pagube insemnate chiar si la icre.
Mierla de parau (Cinclus cinclus Aquaticus L.) si pescarusul verde (Alcedo athis ispida L.) produc pagube insemnate in randul puietilor. Datorita numarului redus de exemplare din zona montana aceste pasari constituie tptusi un pericol scazut pentru populatiile piscicole.
Exista si specii de pesti care sunt considerati daunatori, dintre acestia putand enumera sglavoaca (Cottus gobio) si boisteanul (Phoxinus phoxinus) care consuma icre si puiet.
In Tabelul 1 sunt prezentati pe scurt cativa parametrii fizico-chimici privind calitatea apei din habitatele populate cu salmonide.
Tabel 1. Determinari fizico-chimice privind calitatea apei, pentru habitatele populate cu salmonide.
|
Nr. Crt. |
Bazinul hidrografic |
Cursul de apa |
Parametrii determinati |
||||
|
O2 dizolvat mg/l |
pH |
Temp ˚C |
Duritate |
||||
|
Totala |
Carbonati |
||||||
|
Olt |
lac Bradisor | ||||||
|
Bega-Timis-Nera |
Cerna | ||||||
|
Bega-Timis-Nera |
Nera | ||||||
|
Olt |
Lotru | ||||||
|
Olt |
Latorita | ||||||
|
Jiu |
Tismanita | ||||||
|
Dambovita-Arges-Vedea |
Dambovita | ||||||
|
Dambovita-Arges-Vedea |
lac Vidraru | ||||||
|
Dambovita-Arges-Vedea |
lac Pecineag | ||||||
|
Mures |
Gurghiu | ||||||
|
Ialomita |
Basca Mica | ||||||
Valorile parametrilor determinati se incadreaza in limitele specifice habitatelor populate cu salmonide, duritatea fiind influentata puternic de perioada in care se fac determinarile (anotimp, ploi recente, etc.) si de substratul cursului de apa.
FILOGENIA SALMONIDELOR
In ciuda importantei salmonidelor, istoria lor evolutiva a constituit pentru o lunga perioada de timp un subiect de disputa. Studiile filogenetice anterioare la nivel de gen si specie au oferit informatii importante despre relatiile existente intre reprezentantii familiei Salmonidae, dar mai raman inca destule intrebari, in special vizand speciile genurilor Oncorhynchus si Salvelinus. Speciile de salmonide actuale provin din trei mari linii: Coregoninae, Thymallinae si Salmoninae. In general este acceptat faptul ca toate cele trei linii formeaza un grup monofiletic.
Familia Salmonidae a aparut in Eocenul Mijlociu, reprezentativa fiind specia Eosalmo driftwoodensis. Aceasta specie fosila era destul de asemanatoare cu specia de lipan actuala si poseda o serie de caractere comune liniile actuale Salmoninae, Coregoninae si Thymallinae. Astfel, Eosalmo driftwoodensis reprezinta un salmonid arhaic si constituie un stadiu foarte important in evolutia familiei. Cea mai veche fosila apartinand genului Salvelinus dateaza de acum 10 milioane de ani. Urmatoarele specii fosile identificate dateaza din Miocenul Superior (acum aproximativ 7 milioane de ani). O parte dintre aceste specii fosile par a fi foarte apropiate de genul Oncorhynchus actual, sustinand ipoteza conform careia genul Oncorhynchus a aparut inainte de Pliocen De asemenea, segregarea dintre Oncorhynchus si Salmo s-a petrecut inaintea acestei ere geologice. Speciatia la nivelul genului Oncorhynchus a fost examinata de o perioada indelungata si cu toate acestea, nici la ora actuala nu s-a formulat o concluzie cu privire la interrelatiile existente intre membrii acestui gen.
La fel de controversata este si originea salmonidelor in apa dulce, respectiv apa sarata, existand sustinatori ai ambelor ipoteze. Originea in apa dulce a salmonidelor este sustinuta de faptul ca speciile de salmonide mai primitive, cum ar fi Thymalinae sau Coregoninae, care au aparut inaintea Salmoninaelor, sunt si la ora actuala specii rezidente de apa dulce. Tchernavin (1939) a sugerat ca salmonidele ancestrale erau "pesti de dimensiuni mici, viu colorati care traiau in ape curgatore sau lacuri reci din emisfera nordica". Folosind cursurile de apa dulce, acesti pesti s-au raspandit pe o arie extinsa. Diversitatea ecologica a regiunilor in care s-au raspandit a favorizat formarea numeroaselor specii (Neave, 1958).
Salmonidele prezinta urmatoarea clasificare taxonomica:
Increngatura - Vertebrate
Supraclasa - Pisces
Clasa - Osteichthyes
Subclasa - Actinopterygii
Supraordinul - Teleostei
Ordinul - Salmoniformes
Familia - Salmonidae
Subfamilia - Coregoninae
- Salmoninae
- Thymalinae
Salmonidele apartin clasei Osteichtyes (Huxley 1880), subclasa Actinopterygii (Cope 1887), subdiviziunea Teleostei.
Ordinul Salmoniformes are o singura familie - Salmonidae (Regan 1914) si cuprinde 3 subfamilii: i. Corenidae - cu 10 specii si 3 genuri.
ii. Salmonide - cu 25 specii, cuprinse in 5 genuri.
iii. Thymallidae - cuprinde 4 specii intr-un singur gen.
Initial, filogenia salmonidelor a fost stabilita in urma unor studii de morfologie externa sau dupa caracterele anatomice. In ciuda numarului mare de studii de filogenie clasica, relatiile filogenetice intre anumite specii ale acestui grup de pesti au ramas neclarificate. Cele 66 de specii de salmonide sunt reunite intr-o singura familie - Salmonidae, ce cuprinde trei subfamilii Coregoninae, Thymallinae si Salmoninae. Cea mai numeroasa dintre aceastea, subfamilia Salmoninae include 5 genuri - Hucho, Salmo, Oncorhynchus, Brachymystax si Salvelinus, cu o larga distributie in emisfera nordica.
Putem, deci afirma relatiile filogenetice intre speciile de salmonide sunt complexe si destul de dificil de stabilit. Phillips & Oakley (1997) au considerat ca motivele pentru care precizarea filogeniei salmonidelor este dificila sunt urmatoarele: (1) acesti pesti au suferit o radiatie adaptativa rapida consecutiva tetraploidizarii acum 50-100 milioane de ani; (2) hibridizarea si introgresia reprezinta fenomene comune in cadrul acestui grup de pesti; (3) prin recolonizarea lacurilor rezultate din galciatie in ultimii 10000 de ani a rezultat acumularea unor ecotipuri sau morfotipuri simpatrice (care ocupa acelasi areal sau ale caror areale se suprapun) cu diferite grade de izolare reproductiva in apele din emisfera nordica.
Studiile filogenetice anterioare la nivel de gen si specie au oferit informatii importante despre relatiile existente intre reprezentantii familiei Salmonidae, dar mai raman inca destule intrebari, in special vizand speciile genului Oncorhynchus si Salvelinus.
Speciile de salmonide din Romania au fost carcaterizate din punct de vedere taxonomic prin metode clasice (analize biometrice si morfologice) de Banarescu et al., 1964. Totusi, analiza taxonomica a ajuns in impas datorita imposibilitatii de a determina morfometric diferentele dintre speciile de pesti apropiate si se impune o caracterizare filogenetica a speciilor utilizand criterii moleculare. Tinand seama de importanta economica si stiintifica a acestor specii, este absolut necesara realizarea la salmonide a unor studii de filogenie moleculara bazate pe analiza ADN, luand in considerare si datele clasice de filogenie si sistematica de inalta valoare stiintifica.
Filogenia moleculara a salmonidelor
Filogenia reprezinta o ramura a biologiei care se ocupa cu identificarea si intelegerea relatiilor evolutive existente intre diferiti taxoni, atat actuali, cat si extincti. Teoria evolutiei sustine ca asemanarile intre indivizi sau specii sunt mostenite de la cel mai recent descendent comun. Ca si metodele clasice, metodele de filogenie moleculara se bazeaza pe ipoteza ca asemanarile dintre doua organisme sunt cu atat mai mari cu cat gradul de inrudire este mai mare. In filogenia moleculara, analiza nu se mai face in functie de trasaturi morfologice, ci prin comparare de secvente genice.
Metodele comparative morfologice folosite in stabilirea liniilor filogenetice au fost foarte utile in studii asupra evolutiei, atunci cand nu existau verigi de legatura intre fosilele descoperite. Oricum, aceste metode nu pot furniza o scala a evolutiei corelata cu timpul. Progresul biologiei moleculare din ultimele decenii a furnizat noi metode de studiu asupra evolutiei. Baza acestor metode o constituie gradul mare de stabilitate al secventelor de ADN. Modificarile evolutive in structura secventelor de ADN sunt atat de lente, incat pot furniza informatii detaliate asupra originii si istoriei evolutiei lor.
Din momentul in care, secventele care intra in structura genelor au fost translate in secvente proteice cu ajutorul codului genetic, modificarile evolutive ale aminoacizilor in cadrul proteinelor pot furniza si ele informatii referitoare la procesele si scala aproximativa a timpului evolutiv. Estimarea timpului evolutiv prin aceste metode se bazeaza pe descoperirea potrivit careia, rata de substitutie a aminoacizilor pe an pe site intr-o anumita proteina se produce constant pentru toate organismele.
Stabilirea relatiilor filogenetice pe baza caracterelor moleculare a cunoscut un real progres o data cu aparitia tehnicilor moleculare moderne. Initial, au fost disponibile numai secventele proteice, dar pe masura ce tehnologia a avansat a fost posibila si obtinerea secventelor ADN, tehnica de secventiere constituind in prezent o tehnica de rutina. Atat secventele ADN, cat si secventele proteice sunt folosite pentru estimarea relatiilor filogenetice si a momentului cand a avut loc o evolutie divergenta intre diferiti taxoni. De obicei, secventele ADN sunt utile pentru caracterizarea unor evenimente mai recente din punct de vedere evolutiv. Lungimea secventei utilizate este un factor limitativ. Lungimea medie a unei gene de aproximativ 1000 de nucleotide nu este suficienta pentru stabilirea corecta a relatiilor filogenetice intre diferiti taxoni, de aceea in studiile de filogenie moleculara se recomanda folosirea mai multor gene sau fragmente genice ca markeri filogenetici (Hedges 2002). Un marker filogenetic reprezinta un fragment de ADN codant sau necodant care este folosit in reconstructia profilului filogenetic al unui grup de organisme (taxoni).
Compararea secventelor ADN a anumitor gene de la organisme (specii) diferite poate da informatii utile despre relatiile dintre acele organisme (specii), care altfel nu pot fi obtinute din datele morfologice. Deorece genoamele evolueaza prin acumularea graduala de mutatii, diferentele dintre nucleotidele genoamelor de la organisme diferite informeaza despre cat de recent acestea au avut un ancestor comun. Astfel, doua specii a caror divergenta s-a petrecut in trecutul apropiat prezinta mai putine diferente de secvente genice decat doua specii cu un ancestor comun mai vechi.
O cerinta obligatorie a evolutiei este variatia genetica care rezulta din mutatiile ce apar in timp la nivelul genelor. De fiecare data cand are loc diviziunea celulara, rezulta o copie completa a genomului celulei, proces numit si replicarea ADN. Acest proces trebuie sa fie cat mai exact cu putinta, dar inevitabil, se produc si mutatii. Majoritatea mutatiilor de acest tip nu au nici un efect asupra functiei genelor si se numesc mutatii silentioase. Mutatiile silentioase au rol evolutiv si frecventa aleatoare cu care se produc reprezinta un factor important in evolutie si filogenie. Tinand cont de aceste aspecte, se poate afirma ca, de fapt, filogenia moleculara isi propune sa determine frecventa si tipul modificarilor care apar in structura ADN sau a proteinelor si sa reconstruiasca istoria evolutiva a genelor si a organismelor.
Analiza filogenetica care se bazeaza pe date moleculare creste precizia determinarilor comparativ cu datele morfometrice, desi nici una dintre metodele moleculare nu este complet lipsita de probleme datorita duplicatiilor, deletiilor sau a transferului de gene pe orizontala. Toate acestea pot crea dificultati reale in interpretarea datelor.
Reconstructia profilului filogenetic utilizand markeri nucleari este dificil de realizat, datorita recombinarii meiotice si modului de mostenire diploid al ADN nuclear. Datorita avantajelor pe care le prezinta (mostenirea pe linie materna, rata de evolutie rapida, un nivel de variabilitate si o rata a mutatiilor crescuta), markerii mitocondriali sunt folositi cu succes in studiile de sistematica la diferite nivele taxonomice. Astfel, ADNmt este ideal pentru studii filogenetice si filogeografice (Brown et al., 1979).
Estimarea relatiilor filogenetice pornind de la informatia continuta in secventele ADN se bazeaza pe definirea acelei succesiuni de etape (algoritmi) prin care se poate construi cel mai bun arbore filogenetic. Un arbore filogenetic constituie reprezentarea grafica a filogeniei unui grup de organisme.
Studiile filogenetice in cadrul familiei Salmoninae, bazate pe ADN mitocondrial (ADNmt), au fost initiate de Berg si Ferris care au examinat relatiile filogenetice intre membrii apartinand urmatoarelor genuri Salvelinus, Salmo si Onchorhyncus. Studiile ulterioare (Thomas et al., 1986; Gyllensten et al., 1987; Ginatulina et al., 1988; Thomas et al., 1989; Grewe et al, 1990; Shedlock et al., 1992; Phillips et al., 1995; Kitano et al., 1997; Ohara et al., 1997) s-au axat pe stabilirea relatiilor filogenetice utilizand markeri mitocondriali la speciile apartinand unui singur gen din cele mentionate anterior, celelalte trei fiind utilizate ca outgrup.
Markeri filogenetici mitocondriali
Descrierea morfologica si analiza morfometrica au fost primele tehnici folosite pentru a defini speciile de salmonide, dar metodele de biochimie si biologie moleculara si-au gasit rapid locul intre astfel de studii, inlaturand dubiile legate de o clasificare corecta, mai ales ca la pesti apare frecvent fenomenul hibridizarii interspecifice.
ADN mitocondrial poseda o serie de caracteristici specifice, ca de exemplu organizarea intr-un genom compact, lipsa recombinarii datorita transmiterii uniparentale (mostenire pe linie exclusiv materna), o rata de evolutie rapida. Toate aceste caracteristici recomanda ADNmt ca fiind un marker valoros, cu un real potential de utilizare in studii de genetica a populatiilor si filogenie.
Numarul mare de polimorfisme regasit la nivelul secventelor de nucleotide din doua zone cu hipervariabilitate ale regiunii de control mitocondrial necodificatoare pot permite discriminarea intre indivizi sau probe biologice. Totodata, probabilitatea recuperarii ADNmt din probe biologice foarte mici sau degradate este mai mare decat in cazul ADN nuclear deoarece moleculele de ADNmt sunt prezente in mii de copii per celula, comparativ cu ADN nuclear care se gaseste in doua copii per celula.
In plus, ADNmt este mostenit numai pe linie materna, astfel incat, in cazul in care un individ nu este disponibil pentru o comparatie directa cu o proba biologica, orice proba provenita de la genitorul matern, poate constitui o proba de referinta. Datorita recombinarii meiotice si mostenirii diploide a ADN nuclear, reconstituirea unui profil pe baza ADN nuclear chiar si in cazul rudelor de gradul I este destul de greu de realizat. De asemenea, modelul mostenirii pe linie materna a ADNmt poate fi, de asemeni, considerat ca avand unele deficiente. Deoarece toti indivizii, pe linie materna prezinta aceeasi secventa de ADNmt, aceasta nu poate fi considerata un identificator unic. De fapt, indivizi neinruditi aparent, pot avea o origine comuna si necunoscuta, pe linie materna.
Salmonidele sunt pesti cu importanta comerciala mare si cresterea intensiva in conditii de acvacultura a devenit o preocupare insemnata in multe tari. Ca urmare, este importanta cunoasterea caracteristicilor ADNmt pentru identificarea stocurilor, studiul variabilitatii genetice a populatiilor si stabilirea filogeniei moleculare a salmonidelor (Phillips & Oakley, 1997).
In cadrul familiei Salmonidae, secventa completa a ADNmt este cunoscuta si disponibila in bancile de date pentru urmatoarele specii: Salmo salar (Hurst et al., 1999), Salmo trutta trutta, Salvelinus alpinus (Doiron et al., 1999), Salvelinus fontinalis (Doiron et al., 2002), Onchorhyncus mykiss (Zardoya et al., 1995), O. masou, O. kisutch, O. keta, O. nerka, O. clarkii, O. tshawytscha, C. lavaraetus (Miya & Nishida, 2000), Thymallus articus si Thymallus thymallus.
Similar organizarii de la vertebrate, genomul mitocondrial al pestilor contine 37 de gene, dintre care 13 gene care codifica pentru proteine, 22 de gene pentru ARNt si 2 gene pentru ARNr corespunzatoare transcriptilor 12S si 16S (Figura 26). De exemplu, in cazul speciei Salmo salar, pozitia si orientarea genelor mitocondriale si a regiunii de control este similara organizarii observate la alte specii de pesti sau vertebrate superioare. Cu toate ca organizarea genomului mitocondrial la vertebrate este destul de conservata, la unele specii au fost descrise anumite rearanjamente ale genelor (Kumazawa et al., 1996).
In ceea ce priveste marimea exprimata in perechi de baze (pb) a genomului mitocondrial, aceasta este uniforma la speciile de pesti analizate, diferentele fiind doar de cateva zeci de perechi de baze intre speciile de salmonide (de exemplu, la O. mykiss 16642 pb - Zardoya et al., 1995, la S. salar 16665 pb - Hurst et al., 1999, la S. alpinus 16659 pb - Doiron et al., 1999, la C. lavaraetus 16737 pb - Miya & Nishida, 2000).
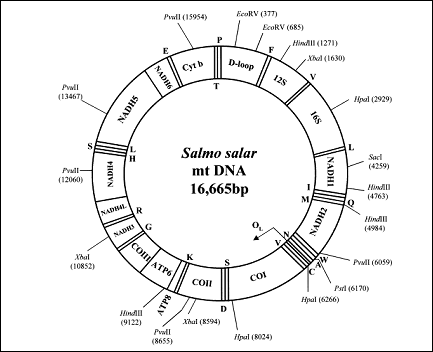
Figura 26. Organizarea genomului mitocondrial la Salmo salar. Sunt indicate enzimele de restrictie folosite pentru clonare in studiul efectuat de Hurst et al., 1999. Numerele reprezinta lungimea in perechi de baze cu originea stabilita arbitrar la capatul 5' al regiunii D-loop. Genele pentru ARNt sunt denumite cu o singura litera, corespunzatoare aminoacidului respectiv.
Caracteristicile ADNmt
Cele doua tipuri de ADN, nuclear si mitocondrial, prezente in celulele vertebratelor, au o origine si o evolutie diferita. ADN mitocondrial prezinta o serie de caracteristici:
este mostenit exclusiv pe linie materna: acest fapt faciliteaza monitorizarea transmiterii diferitelor caractere de-a lungul unor linii evolutive, incepand din stadiile timpurii ale evolutiei. In cazul in care unul dintre indivizii luati in studiu nu are disponibila o proba biologica, orice alta proba care provine de la genitorul matern poate fi utilizata cu succes (San Mauro et al., 2006).
prezinta un nivel crescut de variatie a secventei nucleotidice: ADNmt prezinta un nivel ridicat de variabilitate si o rata crescuta a mutatiilor comparativ cu ADN nuclear. Studiile de la vertebrate au aratat ca divergenta de secventa se acumuleaza mai rapid in ADNmt decat in cel nuclear (Brown, 1985). Acest fenomen a fost atribuit unei rate a mutatiilor mai rapida in ADN mitocondrial, aceasta putand fi rezultatul lipsei mecanismelor de reparare in timpul replicarii (Wilson et al., 1985). De exemplu, numarul mare de polimorfisme intalnite la nivelul a doua regiuni hipervariabile din regiunea de control mitocondrial permite discriminarea intre diferiti indivizi sau diferite probe biologice.
are o rata rapida de evolutie: ADNmt prezinta o rata de evolutie mult mai rapida decat genomul nuclear, fapt ce ii confera o mai mare utilitate in studii de filogenie la specii foarte apropiate.
lipsa recombinarii: o alta caracteristica a ADNmt, general acceptata ca un avantaj real pentru studii de genetica populationala, este lipsa de recombinare (Gillham, 1994; Rokas et al., 2003). Majoritatea animalelor prezinta un tip de reproducere sexuata. Procesul de recombinare lipseste in cazul genelor mitocondriale deoarece genomul mitocondrial este transmis uniparental.
genom compact: aceasta caracteristica permite analiza mai usoara a genomului mitocondrial comparativ cu cel nuclear. Genomul mitocondrial este mai mic, uniform ca marime (15-18 kb la pesti) si astfel, poate fi mai usor de manipulat. In plus, posibilitatea de a obtine ADNmt din probe biologice foarte mici sau chiar degradate este mai mare decat in cazul ADN nuclear, deoarece moleculele de ADNmt se gasesc in mii de copii per celula, in timp ce ADN nuclear prezinta doar doua copii/celula.
Toate aceste proprietati recomanda ADNmt ca fiind un marker foarte util in studii moleculare la pesti, putand fi folosit la:
discriminarea intre specii diferite de salmonide si identificarea hibrizilor;
determinarea relatiilor filogenetice si filogeografice la salmonide;
studii de caracterizare genetica a populatiilor;
programe de acvacultura - de exemplu, in identificarea stocurilor de salmonide si a hibrizilor.
Regiunea de control (D-loop)
Intre genele ARNtPro si ARNtPhe din genomul mitocondrial al vertebratelor se gaseste o regiune necodificatoare care variaza ca lungime (Wolstenholme, 1992). Regiunea mitocondriala de control (D-Loop) reprezinta regiunea reglatoare a genomului mitocondrial al vertebratelor, implicata in initierea replicarii ADN pe catena H, si contine secvente promotoare pentru transcriptia ARN (Clayton, 1982, 1984; Saccone et al., 1991). La mamifere si amfibieni s-a constatat ca aceasta secventa contine semnale necesare initierii replicarii catenei grele (heavy - H) si a fost desemnata ca regiune de control pentru transcrierea ambelor catene mitocondriale, grea (heavy - H) si usoara (light - L) (Shadel & Clayton, 1997). In cazul salmonidelor, regiunea D-loop este asemanatoare ca lungime la speciile studiate si contine 986 pb la O. tshawytscha, 1006 pb la S. salar, 1003 pb la O. mykiss, 964 pb la S. fontinalis, 998 pb la S. alpinus si 1076 pb la C. lavaretus.
Zarodya et al., 1995 a propus la O. mykiss o structura stabila de tipul stem-loop pentru acesta regiune, constand din 11 nucleotide pereche in regiunea stem si o bucla de 17 nucleotide. La Salmo salar a fost propusa o structura secundara asemanatoare, regiunea stem fiind alcatuita din 12 nucleotide pereche, iar regiunea loop din 13 nucleotide. Secventa 5'-GCCGG-3' este conservata in cazul vertebrtatelor, fiind identificata atat la om, cat si la unele specii de salmonide la baza regiunii stem (Figura 27).
Secventa structurii stem este conservata la vertebrate, in timp ce secventa structurii loop prezinta o mai mare variabilitate. In cazul mamiferelor, prin secventierea regiunii loop s-a observat ca aceasta prezinta o secventa bogata in timina necesara initierii replicarii catenei L (Taanman, 1999).
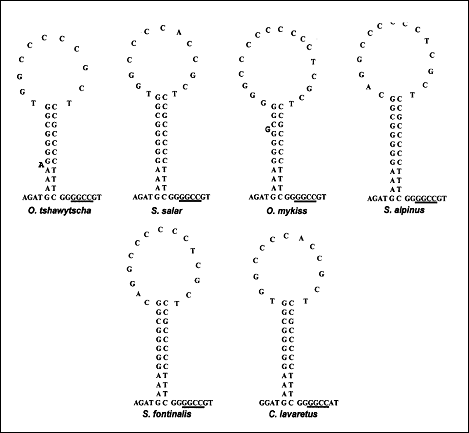
Figura 27. Structura secundara pentru originea replicarii catenei L la 6 specii de salmonide.
In cazul mai multor specii de salmonide, structura loop a regiunii de control este bogata in citozina. Astfel, se pare ca initierea replicarii catenei L nu este conditionata de prezenta timinei, cum s-a crezut initial (Taanman, 1999), ci de prezenta unui lant polipirimidinic. Cateva secvente ale regiunii D-loop prezinta un anumit grad de variatie intre populatiile aceleiasi specii, dar variabilitatea nu este uniforma si regiunea de control mitocondriala prezinta anumite zone bine conservate in seria vertebratelor (Brown et al., 1985; Sbisa et al., 1997). Regiunea D-loop a fost selectata ca marker pentru diverse studii de filogenie moleculara deoarece este considerata cea mai variabila zona mitocondriala in ceea ce priveste rata de substitutie si marimea zonelor variabile (Bentzen et al., 1988; Hoelzel et al., 1991; Meyer, 1993; Cecconi et al., 1995). De asemenea, o variabilitate ampla a zonei D-loop a fost evidentiata la Salmo trutta din diferite regiuni ale Europei (Bernatchez et al., 1992; Giuffra et al., 1994; Bernatchez & Osinov, 1995; Osinov & Bernatchez, 1996; Apostolidis et al., 1997).
MATERIALE SI METODE
Recoltarea probelor de tesut de la salmonide
1. Se recolteaza o portiune mica de tesut, preferabil din inotatoarea caudala, cu ajutorul unor foarfece sau a unui bisturiu. Persoana care colecteaza probele trebuie sa aiba mainile curate, fara mucus sau solzi si foarfecele/bisturiul trebuie sa fie bine clatit intre doua colectari successive (Figura 28).
Marimea portiunii de inotatoare trebuie sa fie de aproximativ 5 cm2, pentru extragerea ADN din inotatoare fiind necesare aproximativ 50 mg de tesut. Daca inotatoarea este prea mica, se recolteaza o portiune din inotatoarea pelvica. Probele pot fi recoltate din tesut proaspat sau inghetat.
2. Fiecare proba de inotatoare va fi stocata separat in criotuburi sau tuburi Eppendorf de 1,5 ml ce contin aproximativ 1 ml de etanol de concentratie superioara (80-96%). Este important ca intreaga portiune de tesut sa fie complet imersata in alcool si sa nu fie expusa contactului cu aerul (Figura 29). Expunerea la aer poate afecta calitatea ADN ce urmeaza sa fie extras din tesut. Etanolul permite pastrarea tesutului la temperatura camerei si nu este necesar ca recipientul cu proba sa fie refrigerat.
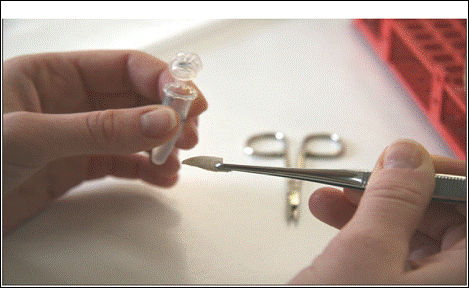
Figura 28. Recoltarea probelor biologice reprezentate de fragmente de inotatoare.

Figura 29. Depozitarea probelor biologice in etanol.
3. Fiecare criotub se eticheteaza corespunzator pentru o identificare ulterioara usoara a probelor. Informatiile continute pe fiecare eticheta se refera la: specia de la care s-a colectat proba, localitatea, numarul probei, data colectarii probei.
4. Depozitarea probelor: probele trebuie ferite de variatii extreme de temperatura, in special daca sunt stocate in etanol, pentru a evita afectarea calitatii ADN.
Pentru extractia ADN cu bune rezultate se pot utiliza si probe reprezentand tesut muscular scheletic sau ficat. Aceste tesuturi dupa recoltare sunt depozitate la -80sC, ulterior fiind folosite pentru extractia ADN. Respectand protocolul de mai sus au fost prelevate probe de salmonide din mai multe specii.
Extractia ADN la salmonide
Izolarea ADN genomic se realizeaza avand la baza un proces de extractie in trei etape. In prima etapa are loc liza celulelor si a nucleelor acestora intr-o solutie continand detergent si EDTA. Eliminarea ARN din extractul de acizi nucleici se realizeaza cu ajutorul unei solutii de ribonucleaza. In etapa urmatoare sunt precipitate proteinele folosind un amestec de solventi organici, in timp ce ADN genomic cu masa moleculara mare ramane in solutie. In final, ADN genomic este concentrat si desalifiat prin precipitare cu etanol absolut. Peletul de ADN precipitat este spalat cu etanol rece 70%, apoi dupa o etapa de centrifugare, urmata de indepartarea etanolului, peletul de ADN este uscat si resuspendat in apa ultrapura sau tampon TE (Tris-EDTA).
Reactivi si aparatura
Fragmente de inotatoare.
EDTA, N-lauroylsarcozinat de sodiu, pronaza, ribonucleaza.
Fenol, cloroform, alcool izoamilic, etanol absolut si 70%, apa ultrapura.
Tuburi de centrifuga sterile de 1,5 ml.
Agitator REAX Top (Heidolph).
Microcentrifuga MiniSpin (Eppendorf).
- Termobloc TDB 120 (Biosan).
Mod de lucru
Ziua I
Intr-un tub Eppi de 2 ml se adauga EDTA (etilen diamin-tetraacetic acid, tetra sodic x 2 H2O), sarcozinat de sodiu si pronaza.
Se adauga tesut in fiecare tub.
Se amesteca usor si se incubeaza 15-16 ore.
Ziua II
Se adauga 10 l ribonucleaza in fiecare tub, se amesteca si se incubeaza la 37sC timp de o ora.
Se adauga 400 µl fenol in fiecare tub. Se agita puternic fiecare tub aproximativ 20 secunde si apoi mult mai usor timp de 10 minute (prin inversia repetata a tubului sau prin vortexare la viteza mica).
Se adauga 400 µl cloroform:alcool izoamilic (24:1) in fiecare tub. Se agita puternic timp de 20 secunde, apoi mult mai usor timp de 10 minute.
Se centrifugheaza tuburile la 13400 rpm timp de 3 minute.
Cu o pipeta automata, se indeparteaza cu atentie stratul apos superior si se transfera intr-un nou tub.
Se repeta pasii 3, 4, 5 pe faza superioara nou transferata.
Se adauga doua volume de etanol 100% rece peste faza apoasa transferata in tub. Se amesteca prin inversia rapida a tubului de 5-6 ori.
Se centrifugheaza si apoi se arunca etanolul prin inversia tubului.
Intr-un interval de maxim 15 minute se decanteaza cu atentie etanolul. Se adauga 1 ml de etanol 70% si se spala pentru cel putin o ora prin agitare usoara.
Se centrifugheaza 3 minute la 13400 rpm si apoi se decanteaza etanolul 70% adaugat, indepartand excesul prin inclinarea tubului.
ADN se usuca la 37˚C timp de 10-15 minute si se resuspenda in apa PCR. Se lasa cel putin 24 de ore pentru dizolvare completa la 4˚C.
Puritatea si concentratia probelor ADN se verifica spectrofotometric. Raportul optim A260/A280 este cuprins intre 1,8 si 2. Daca raportul inregistreaza valori sub 1,8, solutia ADN este contaminata cu proteine, iar daca valoarea acestuia depaseste 2, cantitatea de ARN din proba este peste limita admisa si va interfera cu operatiile ulterioare.
In cazul excesului de ARN se pot constata amplificari nespecifice sau hibridizari intre ARN-primeri si ARN-ADN. Atunci cand apare contaminarea se repeta pasii specifici de purificare pana se obtine o valoare care sa fie cuprinsa in domeniul optim. Integritatea ADN a fost verificata prin electroforeza in gel de agaroza 1%.
Desemnarea de primeri specifici pentru amplificarea secventelor ADN de interes filogenetic
Fragmentul ADN care urmeza sa fie amplificat este desemnat prin selectarea primerilor. Primerii reprezinta oligocatene scurte, artificiale de ADN, nu mai lungi de 50 pb (de obicei de 18-25 nucleotide), care marcheaza inceputul si sfarsitul fragmentului ADN ce urmeaza sa fie amplificat. Primerii hibridizeaza la catena ADN formand puncte de inceput si de sfarsit, unde ADN polimeraza se leaga si incepe sinteza unei noi catene ADN.
Alegerea lungimii si temperaturii de hibridizare a primerilor (Tm) depinde de un numar de factori. Temperatura de hibridizare a primerilor este definita ca temperatura la care primerul se va lega pe baza de complementaritate la catena ADN si deasupra careia primerul va disocia de ADN. Temperatura de hibridizare creste cu lungimea primerului. Primerii care sunt prea scurti vor adera in cateva pozitii de-a lungul catenei de ADN ceea ce va determina generarea de copii nespecifice.
Uneori sunt folositi primeri degenerati. Acestia reprezinta de fapt un amestec de primeri similari, dar nu identici. Acest tip de primeri este potrivit daca se doreste amplificarea aceleiasi gene de la organisme diferite, deorece aceste gene sunt similare, dar nu identice. Folosirea primerilor degenerati poate reduce specificitatea amplificarii PCR. Problema poate fi partial rezolvata prin folosirea tehnicii "touchdown PCR".
Din cauza considerentelor mentionate, desemnarea primerilor reprezinta un proces precis, de care depinde cantitatea de produs obtinut:
- Continutul de GC nu trebuie sa fie intre 40-60%.
- Temperatura de melting calculata pentru ambii primeri utilizati in reactie nu trebuie sa difere cu maxim 5sC si temperatura de melting a produsului de amplificare nu trebuie sa difere de cea a primerilor cu maxim 10 sC.
- Temperatura de hibridizare este de obicei cu 5sC mai mica fata de temperatura de topire (Tm) calculata.
- Trebuie evitata formarea dimerilor de primeri.
- Capatul 3' este foarte sensibil si nu trebuie sa fie complementar cu orice alta regiune a altui primer folosit in recatie.
La ora actuala exista programe de calculator care sunt folosite cu succes in desemnarea primerilor.
Desemnarea primerilor specifici pentru amplificarea secventelor D-loop la speciile de salmonide analizate s-a realizat cu ajutorul programului Primer3 (https://frodo.wi.mit.edu/primer3/), urmarind urmatoarele etape:
Secventa de interes a fost descarcata din baza de date NCBI (National Center for Biotechnology Information) si salvata intr-un format specific (Figura 30).
Secventa a fost introdusa in programul Primer3 pastrandu-se setarile recomandate. Se selecteaza <Pick left primer> pentru desemnarea primerului sens, respectiv <Pick right primer> pentru primerul antisens. Pentru alegerea primerilor se selecteaza comanda <Pick Primers> (Figura 31).
Secventele primerului sens, respectiv antisens, generate de programul Primer3 vor fi prezentate intr-o fereastra noua (Figura 32).
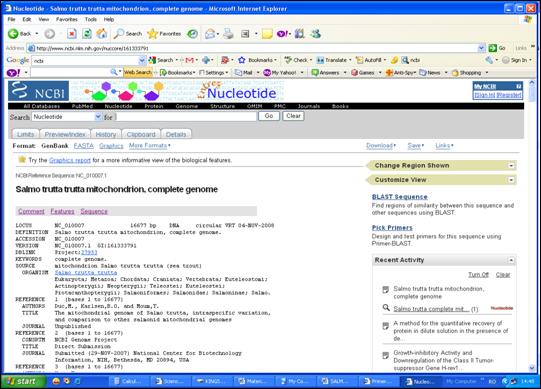
Figura 30. Accesarea secventei nucleotidice de interes din GenBank prin pagina de web www.ncbi.nlm.nih.gov.
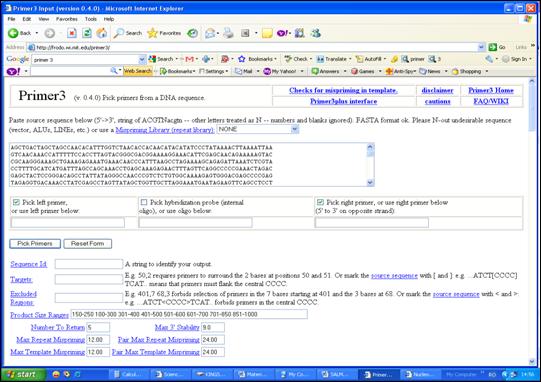
Figura 31. Interfata programului de desemnare a primerilor Primer3.
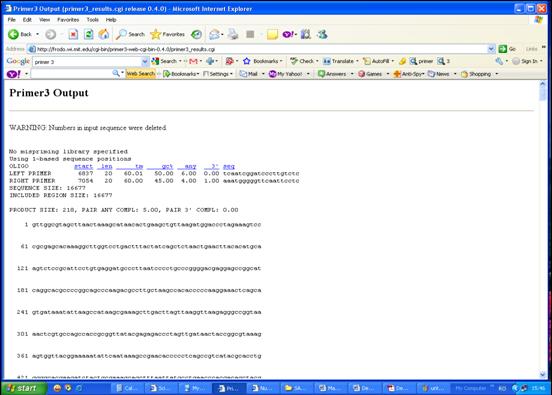
Figura 32. Secventele primerilor obtinuti.
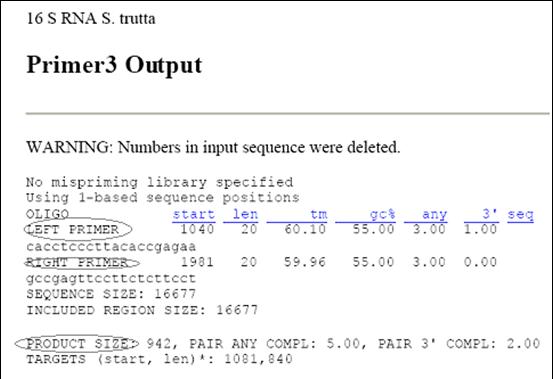
Figura 33. Secventele primerilor generate de programul Primer3.
In noul format generat de Primer3, dupa selectarea comenzii <Pick Primers>, se regasesc urmatoarele informatii: secventa primerilor sens si antisens, situsul de legare a primerilor, temperatura de melting si procentul GC al primerilor, lungimea produsului PCR amplificat cu primerii respectivi (Figura 33).
Secventele primerilor vor fi introduse pentru verificarea identitatii intr-un program din baza de date GenBank: BLAST. Acesta parcurge secventele din banca de date, oprindu-se la fiecare regiune din secventa bancii care are corespondenta cu secventa data. BLAST incearca sa realizeze un aliniament local, fara sa insereze spatii in aceste secvente. Rezultatul obtinut prin analiza BLAST este redat in cateva secunde sub forma unui tabel in care sunt afisate toate procentajele de identitati obtinute (Figurile 34-35).
In cazul in care secventa cautata se regaseste in baza de date, rezultatul afisat de catre aplicatia BLAST il reprezinta genele cu identitatile de secventa afisate in ordine descrescatoare (Figura 36).
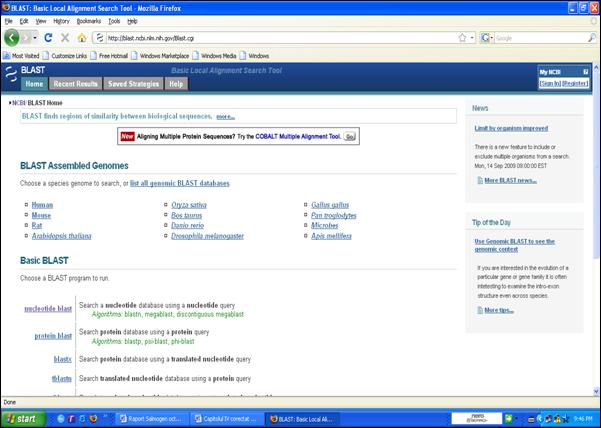
Figura 34. Accesarea programului BLAST prin pagina de web www.ncbi.nlm.nih.gov.
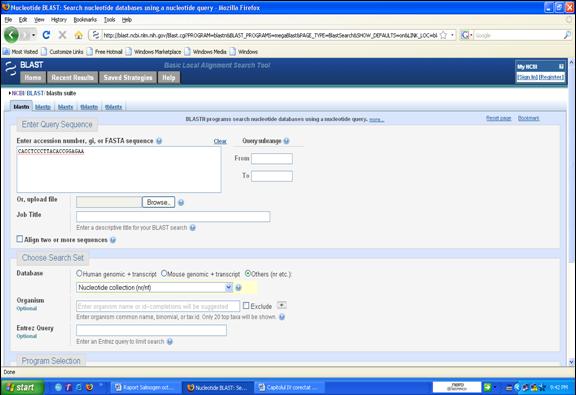
Figura 35. Introducerea secventei de interes.
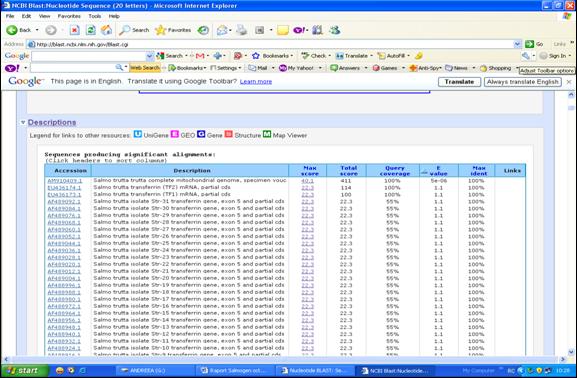
Figura 36. Rezultatul analizei BLAST.
Determinarea temperaturii de hibridizare pentru fiecare set de primeri prin PCR in gradient de temperatura
Reactia de polimerizare in lant (PCR) reprezinta o tehnica de biologie moleculara prin care se realizeaza amplificarea anumitor fragmente din molecula de ADN. Tehnica este utilizata pentru amplificarea unei parti bine definite dintr-o catena ADN. Spre deosebire de procesul de replicare din organsimele vii, prin PCR se pot copia numai fragmente scurte de ADN, de obicei pana la 7-8 kb. Prin anumite metode se pot copia si fragmente mari, pana la 40 kb, ceea ce reprezinta totusi mult mai putin fata de marimea ADN genomic.
Tehnica PCR necesita cateva componente de baza. Aceste componente sunt: matrita ADN, care contine regiunea care urmeaza sa fie amplificata, oligonucleotide complementare sau primeri, care marcheaza inceputul si sfarsitul regiunii ce urmeza a fi amplificata, ADN polimeraza stabila termic, care realizeaza amplificarea propriu-zisa, nucleotidele cu ajutorul carora ADN polimeraza sintetizeaza noul fragment de ADN, tamponul de reactie care confera un mediu chimic corespunzator pentru desfasurarea reactiei.
Reactia PCR consta intr-o serie de 35-45 de cicluri, fiecare fiind alcatuit din trei pasi:
1) dublul helix de ADN trebuie incalzit la 94-96sC pentru separarea catenelor. Acest pas se numeste denaturare. In timpul acestui proces sunt rupte legaturile de hidrogen care unesc cele doua catene ADN. Inainte de primul ciclu, ADN este de obicei denaturat pentru un timp mai extins pentru a fi siguri ca dublele catene ADN s-au separat complet si acum se afla in stare monocatenara.
2) dupa separarea catenelor, temperatura scade pentru a permite primerilor sa se ataseze la monocatena ADN. Acest pas se numeste hibridizare. Temperatura la care se desfasoara acest pas depinde de primeri si este de obicei cu 5sC mai scazuta fata de temperatura lor de melting (45-68 sC). O temperatura de hibridizare eronata poate determina nelegarea primerilor la catena ADN sau legarea la intamplare a primerilor.
3) in final, ADN polimeraza trebuie sa completeze catena lipsa. Acest pas se numeste elongare sau extensie si incepe la locul de anelare a primerului si se desfasoara de-a lungul catenei ADN. Temperatura de elongare depinde de ADN polimeraza si de lungimea fragmentului ADN amplificat. Majoritatea ADN polimerazelor utilizate provin din organisme termofile si functioneaza optim la 70-80sC.
Pentru optimizarea reactiilor de amplificare am realizat, pentru fiecare dintre regiunile D-loop la speciile analizate, reactii de PCR in gradient de temperatura. In cazul acestui tip de reactie, ADN matrita este supus concomitent unor temperaturi diferite de hibridizare a primerilor in scopul stabilirii temperaturii optime care sa permita eliminarea amplificarilor parazite si realizarea cu un randament maxim a reactiei PCR. Totodata, in decursul unei astfel de reactii de optimizare, pot fi variate si intervalele de timp in care sunt parcurse treptele de temperatura, cat si numarul de cicluri de amplificare.
De asemenea, prin aceiasi tehnica au fost optimizate si temperaturile de hibridizare a primerilor pentru amplificarea unei regiuni extinse a genomului mitocondrial al salmonidelor in vederea efectuarii unei reactii PCR-RFLP.
Reactivi si aparatura:
Probe de ADN extrase de la exemplarele ce urmeaza sa fie analizate.
Reactivi PCR:
amestec de deoxi-nucleotide 10 mM din fiecare;
clorura de magneziu 25 mM;
tampon PCR 10X care contine 100 mM TRIS-HCl, 500 mM KCl, pH 8,3;
polimeraza AmpliTaq Gold 5 U/μl;
primeri sens si antisens.
Reactivi pentru electroforeza:
tampon de electroforeza: TAE 1X - TRIS 0,040 M, Acid acetic 0,040 M, EDTA 0,002 M. Se prepara sub forma unei solutii 10X care este apoi diluata;
tamponul probei: albastru de bromfenol 0,3%, glicerol 30% in TAE 1X;
solutie de bromura de etidium 10 mg/ml;
marker de masa moleculara 100 bp (Promega);
agaroza.
Tanc de electroforeza orizontala pentru acizi nucleici Mini-Sub CELL GT.
Sursa de curent PowerPac Basic (Bio-Rad).
Sistem de video documentare BIO-PROFIL (Vilber-Lourmat).
Termociclor iCycler (BioRad).
Termociclor GeneAmp PCR System 9700 (Applied Biosystems).
Microcentrifuga MiniSpin (Eppendorf).
Mod de lucru:
S-a realizat determinarea temperaturii de hibridizare specifice fiecarei perechi de primeri desemnate pentru amplificarea regiunii mitocondriale D-loop si pentru amplificarea unei regiuni mitocondriale extinse prin PCR in gradient de temperatura.
- se amesteca componentele de reactie (Tabel 2); vortexare si centrifugare;
- se adauga 20 ml din amestecul de reactie in cate un tub de PCR de 0,2 μl pentru fiecare proba analizata;
- se adauga 5 ml ADN, diluat corespunzator; vortexare si centrifugare;
- tuburile se aseaza in aparatul de PCR.
Tabel 2. Componentele amestecului PCR.
|
Componenta |
Volum (μl) |
|
PCR Buffer 10X | |
|
MgCl2 25 mM | |
|
dNTP 10 mM | |
|
Primer forward | |
|
Primer reverse | |
|
AmpliTaq Gold Polimeraza | |
|
Apa PCR |
Pentru realizarea reactiei PCR in gradient de temperatura s-a programat aparatul de PCR respectand parametri prezentati in Tabelele 3 si 4.
La terminarea reactiei PCR se efectueaza o electroforeza in gel de agaroza in vederea vizualizarii produsilor de amplificare.
Initial se prepara gelul de agaroza astfel:
- se izoleaza tavita aparatului de electroforeza cu banda adeziva;
- se prepara un volum de 40 ml de agaroza 2% in tampon TAE 1X;
- dupa dizolvarea completa a agarozei la cald se adauga 2,3 μl bromura de etidium;
- se monteaza pieptenul si apoi se toarna gelul in tavita de electroforeza;
- se lasa aproximativ 40 de minute la temperatura camerei pentru a se solidifica, iar gelul se introduce in tancul de electroforeza.
Produsii de amplificare se amesteca cu tamponul probei si se incarca in godeuri cu o pipeta automata. Se conecteaza tancul de electroforeza la sursa de curent si se porneste electroforeza. Migrarea se desfasoara la 80V constant si la temperatura camerei, timp de 40 de minute. La finalul migrarii, vizualizarea benzilor de ADN se realizeaza pe un transiluminator UV. In functie de profilul electroforetic obtinut s-a ales temperatura de hibridizare optima pentru a permite analiza genelor luate in studiu.
|
Specia |
Aparat de PCR |
Timpii si temperaturile de incubare |
|||||
|
O. mykiss |
Etapa initiala |
40 Cicluri |
Extensie finala | ||||
|
Denaturare |
Anelare |
Extindere |
|||||
|
iCycler (BioRad) |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
30 secunde 51 - 61 oC |
1 minut 72oC |
10 min. 72oC 1 ciclu |
4oC |
|
|
Specia |
Aparat de PCR |
Timpii si temperaturile de incubare |
|||||
|
S. trutta |
Etapa initiala |
40 Cicluri |
Extensie finala | ||||
|
Denaturare |
Anelare |
Extindere |
|||||
|
Palm Cycler (Corbett) |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
40 secunde 51 - 61 oC |
90 secunde 72oC |
20 min. 72oC 1 ciclu |
4oC |
|
|
Specia |
Aparat de PCR |
Timpii si temperaturile de incubare |
|||||
|
S. fontinalis |
Etapa initiala |
40 Cicluri |
Extensie finala | ||||
|
Denaturare |
Anelare |
Extindere |
|||||
|
Palm Cycler (Corbett) |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
40 secunde 51 - 61 oC |
90 secunde 72oC |
20 min. 72oC 1 ciclu |
4oC |
|
|
Specia |
Aparat de PCR |
Timpii si temperaturile de incubare |
|||||
|
T. thymallus |
Etapa initiala |
40 Cicluri |
Extensie finala | ||||
|
Denaturare |
Anelare |
Extindere |
|||||
|
Palm Cycler (Corbett) |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
30 secunde 51 - 61 oC |
1 minut 72oC |
10 min. 72oC 1 ciclu |
4oC |
|
|
Specia |
Timpii si temperaturile de incubare |
|||||
|
Etapa initiala |
45 de cicluri |
Extensie finala |
Etapa finala 4oC |
|||
|
Denaturare |
Anelare |
Extindere |
||||
|
Salmo trutta |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
45 secunde 51-62oC |
2 minute 72oC |
20 minute 72oC 1 ciclu |
|
|
Salmo trutta labrax |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
45 secunde 51-62oC |
2 minute 72oC |
20 minute 72oC 1 ciclu |
4oC |
|
Salvelinus fontinalis |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
45 secunde 51-62oC |
2 minute 72oC |
20 minute 72oC 1 ciclu |
4oC |
|
Hucho hucho |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
45 secunde 51-62oC |
2 minute 72oC |
20 minute 72oC 1 ciclu |
4oC |
|
Onchorhyncus mykiss |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
45 secunde 51-62oC |
2 minute 72oC |
20 minute 72oC 1 ciclu |
4oC |
|
Thymallus thymallus |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
45 secunde 51-62oC |
2 minute 72oC |
20 minute 72oC 1 ciclu |
4oC |
Amplificarea regiunii D-loop mitocondriale la salmonide
Pentru analiza regiunii D-loop mitocondriale si stabilirea relatiilor filogenetice existente intre speciile de salmonide din Romania se realizeaza mai intai o reactie de amplificare prin metoda PCR a ADN extras anterior. Dupa amplificare fragmentele obtinute sunt supuse unei elecroforeze in gel de agaroza 2%. Estimarea marimii fragmentelor amplificate se face prin migrarea concomitenta a unui marker de masa moleculara.
Reactivi si aparatura:
Probe de ADN extrase de la exemplarele ce urmeaza sa fie analizate.
Reactivi PCR:
o amestec de deoxi-nucleotide, 10 mM din fiecare;
o clorura de magneziu 25 mM;
o tampon PCR 10X care contine 100 mM TRIS-HCl, 500 mM KCl, pH 8,3;
o ADN polimeraza AmpliTaq Gold;
o apa ultrapura;
o primeri sens si antisens
Reactivi pentru electroforeza:
o tampon de electroforeza: TAE 1X - TRIS 0,040 M, acid acetic 0,040 M, EDTA 0,002 M. Se prepara sub forma unei solutii 10X care este apoi diluata;
o tamponul probei: albastru de bromfenol 0,3%, glicerol 30% in TAE 1X;
o solutie de bromura de etidium 0,5 mg/ml;
o marker de masa moleculara DNA Ladder 100 bp (Promega);
o agaroza.
Tanc de electroforeza orizontala pentru acizi nucleici Mini-Sub CELL GT si sursa de curent PowerPac Basic (Bio-Rad).
Sistem de video documentare BIO-PROFIL (Vilber-Lourmat).
Plita cu incalzire si agitare magnetica MR 3001 (Heidolph).
Formamida deionizata.
Termociclor GeneAmp PCR System 9700 (Applied Biosystems).
Analizator genetic automat ABI Prism 3130 (Applied Biosystems).
Microcentrifuga MiniSpin (Eppendorf).
Agitator REAX Top (Heidolph).
Termobloc TDB 120 (Biosan).
Mod de lucru
Pentru fiecare exemplar analizat reactia PCR a fost realizata in modul urmator:
se adauga reactivii prezentati in Tabelul 5 pentru a obtine amestecul de reactie; vortexare si centrifugare;
se adauga 20 ml din amestecul de reactie in cate un tub de PCR;
se adauga 5 ml din ADN diluat corespunzator de la fiecare exemplar;
vortexare si centrifugare;
tuburile se aseaza in aparatul de PCR.
Pentru realizarea reactiei PCR:
se programeaza aparatul de PCR la parametrii indicati in Tabelul 6.
amplificare PCR;
produsii obtinuti sunt pastrati la 2 - 6 C pana la analiza.
Produsii obtinuti sunt supusi unei electroforeze orizontale, in gel de agaroza 2%, in sistem submers (tampon TAE 1X). Vizualizarea gelului se va face in lumina UV.
Tabel 5. Componentele si volumele amestecului de PCR.
|
Componente |
Volum (μl) |
|
Tampon PCR | |
|
Clorura de magneziu | |
|
Amestec de nucleotide | |
|
ADN polimeraza AmpliTaq Gold | |
|
Primer F | |
|
Primer R | |
|
Apa ultrapura | |
|
Volum total |
|
Specia |
Aparat de PCR |
Timpii si temperaturile de incubare |
|||||||||||||
|
O. mykiss |
Etapa initiala |
40 Cicluri |
Extensie finala | ||||||||||||
|
Denaturare |
Anelare |
Extindere |
|||||||||||||
|
GeneAmp PCR System 9700 |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
30 secunde 58oC |
1 minut 72oC |
10 min. 72oC 1 ciclu |
4oC |
|||||||||
|
Specia |
Aparat de PCR |
Timpii si temperaturile de incubare |
Etapa initiala |
40 Cicluri |
Extensie finala |
|
|||||||||
|
Denaturare |
Anelare |
Extindere |
|||||||||||||
|
GeneAmp PCR System 9700 |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
40 secunde 57oC |
1 minut si 30 secunde 72oC |
20 min. 72oC 1 ciclu |
4oC |
|
||||||||
|
Specia |
Aparat de PCR |
Timpii si temperaturile de incubare |
Etapa initiala |
40 Cicluri |
Extensie finala |
|
|||||||||
|
Denaturare |
Anelare |
Extindere |
|||||||||||||
|
GeneAmp PCR System 9700 |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
40 secunde 56oC |
1 minut si 30 secunde 72oC |
20 min. 72oC 1 ciclu |
4oC |
|
||||||||
|
Specia |
Aparat de PCR |
Timpii si temperaturile de incubare |
Etapa initiala |
40 Cicluri |
Extensie finala |
|
|||||||||
|
Denaturare |
Anelare |
Extindere |
|||||||||||||
|
GeneAmp PCR System 9700 |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
30 secunde 58oC |
1 minut 72oC |
10 min. 72oC 1 ciclu |
4oC |
|
||||||||
Tehnica PCR-RFLP
Principiul metodei
In cazul salmonidelor tehnica PCR-RFLP este utilizata cu succes in realizarea unei diferentieri interspecifice pe baze moleculare. Astfel, este amplificata prin tehnica PCR o regiune ampla (aproximativ 2300 pb) de la nivelul genomului mitocondrial, iar ampliconii obtinuti sunt digerati cu ajutorul unor enzime de restrictie specifice. Amprenta fragmentelor rezultate in urma digestiei difera de la o specie la alta furnizand astfel o metoda moleculara eficienta de diferentiere intre speciile de salmonide.
Material biologic: probe de ADN genomic extrase de la exemplare de salmonide provenind din specii diferite.
Reactivi si aparatura pentru reactia de amplificare: i) amestec de deoxinucleotide, 10 mM din fiecare; ii) clorura de magneziu 25 mM; iii) tampon PCR 10X care contine 100 mM TRIS-HCl, 500 mM KCl, pH 8,3; iv) ADN polimeraza 5 U/μl; v) apa ultrapura; vi) primer sens, 20 μM si primer antisens, 20 μM; vii) aparat PCR; viii) minicentrifuga; ix) agitator.
Reactivi pentru reactia de restrictie: i) tampon de restrictie: 60 mM TRIS-HCl, 1 mM NaCl, 60 mM MgCl2, 10 mM DTT (ditiotreitol) pH 7,5; ii) albumina serica bovina (BSA) acetilata 10 μg/μl; iii) endonucleaza de restrictie Hinf I 10 U/μl; iv) minicentrifuga; v) agitator; vi) termobloc.
Reactivi si aparatura pentru electroforeza: i) tampon de electroforeza TAE 1X - TRIS 40 mM, acid acetic 40 mM, EDTA 2 mM care se prepara ca solutie 10X si este diluat inainte de folosire; ii) tamponul probei care contine albastru de bromfenol 0,3% si glicerol 30% in TAE 1X; iii) solutie de bromura de etidiu 10 mg/ml; iv) marker de masa moleculara; v) agaroza; vi) tanc de electroforeza orizontala pentru acizi nucleici; vii) sursa de curent; viii) transiluminator UV; ix) plita cu incalzire si agitare magnetica.
Mod de lucru
Amplificarea prin PCR
1. Reactivii prezentati in tabelul 7 se adauga intr-un tub de minicentrifuga pentru a obtine amestecul de reactie. Se agita si se centrifugheaza usor.
2. Se adauga 20 ml din amestecul de reactie in cate un tub de centrifuga de 200 μl pentru fiecare exemplar testat.
3. Se adauga 5 ml ADN extras de la fiecare exemplar de testat, diluat corespunzator (circa 50 ng/reactie). Se agita si se centrifugheaza usor.
4. Tuburile se aseaza in aparatul PCR si acesta este setat in conformitate cu programul prezentat in tabelul 8.
Tabel 7. Reactivi si cantitati necesare realizarii reactiei PCR.
|
Componente |
Volum (μl) |
|
Tampon PCR 10X | |
|
MgCl2 | |
|
dNTP | |
|
ADN polimeraza | |
|
Primer sens | |
|
Primer antisens | |
|
Apa ultrapura | |
|
Volum total |
|
Specia |
Timpii si temperaturile de incubare |
|||||
|
Etapa initiala |
45 de cicluri |
Extensie finala |
Etapa finala 4oC |
|||
|
Denaturare |
Anelare |
Extindere |
||||
|
Salmo trutta |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
45 secunde 56oC |
2 minute 72oC |
20 minute 72oC 1 ciclu |
|
|
Salmo trutta labrax |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
45 secunde 56oC |
2 minute 72oC |
20 minute 72oC 1 ciclu |
4oC |
|
Salvelinus fontinalis |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
45 secunde 53oC |
2 minute 72oC |
20 minute 72oC 1 ciclu |
4oC |
|
Hucho hucho |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
45 secunde 56oC |
2 minute 72oC |
20 minute 72oC 1 ciclu |
4oC |
|
Onchorhyncus mykiss |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
45 secunde 56oC |
2 minute 72oC |
20 minute 72oC 1 ciclu |
4oC |
|
Thymallus thymallus |
10 min. 95oC 1 ciclu |
30 secunde 95oC |
45 secunde 58oC |
2 minute 72oC |
20 minute 72oC 1 ciclu |
4oC |
Electroforeza in gel de agaroza in vederea vizualizarii produsilor de amplificare
Dupa terminarea reactiei PCR se realizeaza o electroforeza in gel de agaroza pentru vizualizarea produsilor de amplificare. Produsii de amplificare se amesteca cu tamponul probei si se incarca in godeuri. Se conecteaza tancul la sursa de curent si se porneste electroforeza. Migrarea se desfasoara la tensiune constanta si la temperatura camerei, timp de 40-50 de minute, in functie de marimea fragmentelor amplificate.
La finalul migrarii, vizualizarea benzilor de ADN se realizeaza cu ajutorul unui transiluminator UV.
Reactia de restrictie
1. Se adauga reactivii prezentati in Tabelul 9 intr-un tub de minicentrifuga pentru a obtine amestecul de restrictie. Se agita si se centrifugheaza.
2. Digestia se desfasoara la 37sC, timp de 3 ore.
Tabel 9. Componentele si cantitatile necesare efectuarii reactiei de restrictie.
|
Componente |
Volum (μl) |
|
Tampon de restrictie 10X | |
|
BSA acetilata | |
|
Produs PCR | |
|
Hinf I | |
|
Apa ultrapura | |
|
Volum total |
Electroforeza in gel de agaroza in vederea vizualizarii produsilor de restrictie
Dupa terminarea reactiei de restrictie se realizeaza o electroforeza in gel de agaroza pentru vizualizarea produsilor. Produsii de restrictie se amesteca cu tamponul probei si se incarca in godeuri. Se conecteaza tancul la sursa de curent si se porneste electroforeza. Migrarea se desfasoara la tensiune constanta si la temperatura camerei, timp de 40-60 de minute, in functie de marimea fragmentelor. La finalul migrarii, vizualizarea benzilor de ADN se realizeaza cu ajutorul unui transiluminator UV.
Secventierea ADN
Secventierea ADN reprezinta procesul prin care este determinata compozitia si ordinea nucleotidelor intr-un fragment ADN. Metoda de secventiere dideoxi sau enzimatica, original dezvoltata de Sanger si colaboratorii sai, utilizeaza ADN polimeraza pentru a sintetiza o copie complementara unei singure catene din proba de ADN. In afara de ADN si ADN polimeraza, in amestecul de reactie se adauga un primer care recunoaste pe baza de specificitate catena ADN, cele patru deoxinucleotide (dATP, dGTP, dCTP, dTTP) in proportii egale si cele patru dideoxinucleotide (ddATP, ddGTP, ddCTP, ddTTP) marcate fluorescent. Dideoxinucleotidele sunt nucleotide sintetice carora le lipseste gruparea -OH de la nivelul atomului C3'. Raportul ddNTP/dNTP din fiecare reactie este ajustat astfel incat fiecare nucleotida sa participe la reactie. Elongarea catenei ADN se va produce pana cand ADN polimeraza va insera aleator un dideoxinucleotid in locul unui deoxinucleotid. Extensia catenei este stopata deoarece lipseste gruparea C3'-OH pentru atasarea urmatorului nucleotid. In final, rezulta un amestec de fragmente cu capatul 5` determinat de primer si capatul 3`terminat cu un ddNTP marcat fluorescent. Fragmentele rezultate vor diferi in final in lungime doar la nivelul unei perechi de baze.
Tehnica de secventiere ADN este bazata pe tehnica de electroforeza capilara de inalta rezolutie. Aceasta este capabila de a separa si diferentia cu o rezolutie de pana la o singura pereche de baze. Fragmentele rezultate sunt separate in functie de dimensiuni de la cel mai lung la cel mai scurt.
Fragmentele ADN obtinute prin PCR au fost secventiate pentru a permite analiza regiunii D-loop mitocondriale pentru speciile de salmonide analizate. Initial, ampliconii rezultati in urma reactiei PCR au fost purificati. Daca nu exista benzi suplimentare care sa denote amplificari nespecifice, purificarea ampliconilor se realizeaza direct din produsul PCR utilizand kitul Wizard PCR Preps DNA Purification System (Promega).
Purificarea directa a ADN
Se transfera produsii PCR in tuburi Eppendorf de 1,5 mL.
Se aduga 100 μL Direct Purification Buffer. Se vortexeaza si se centrifugheaza.
Se adauga 1 ml de rasina ce a fost incubata in prealabil 10 minute la 37˚C peste amestecul respectiv si se amesteca bine 20 - 30 de secunde prin vortexare rapida.
Se pregateste o coloana Wizard pentru fiecare amplicon. Se pipeteaza amestecul rasina-ADN in coloana si apoi aceasta se conecteaza la pompa de vid.
Se indeparteaza pompa de vid si se spala coloana cu 2 ml izopropanol 80 %. Se reconecteaza coloana la vid.
Se usuca rasina prin mentinerea la vid timp de 30 de secunde. Coloana se indeparteaza si se transfera intr-un tub de centrifuga nou.
Se centrifugheaza 2 minute la 10000g pentru a indeparta izopropanolul.
Coloana se transfera intr-un alt tub de centrifuga curat, se adauga 50 μl apa PCR si se incubeaza 1 minut la temperatura camerei.
Se centrifugheaza 20 de secunde la 10000 g pentru a elua fragmentele de ADN. ADN purificat poate fi pastrat la 4 0C sau la - 20 0C (pentru un timp indelungat).
Purificarea din gel de agaroza a ADN
1. Se adauga 1 ml de rasina care a fost incubata in prealabil 10 minute la 37˚C peste agaroza topita si se amesteca bine timp de 20 de secunde prin inversarea tubului.
2. In continuare se urmeaza pasii 5-11 din protocolul de mai sus.
Calitatea si concentratia ADN obtinut dupa reactia PCR si purificare a fost evaluata prin determinarea absorbantei la 260/280 nm.
Fragmentele PCR purificate si cuantificate spectrofotometric au fost supuse secventierii folosindu-se kitul BigDye Terminator Cycle Sequecing Ready Reaction v3.1 (Applied Biosystems). Secventierea presupune trei etape: i) amplificarea PCR cu un singur primer (sens sau antisens), ii) purificarea produsilor obtinuti in urma reactiei de amplificare, iii) incarcarea si rularea probelor pe secventiatorul ABI Prism 3130 Genetic Analyzer (Applied Biosystems). Pentru realizarea reactiilor PCR se programeaza aparatul programul din Tabelul 10. Produsii PCR obtinuti au fost precipitati folosind kitul de purificare BigDye X-Terminator (Applied Biosystems).
Tabel 10. Schema de realizare a reactiei PCR pentru secventiere.
|
Denaturare initiala |
Cicluri | |||
|
Denaturare |
Anelare |
Extensie |
||
|
1 ciclu 96oC - 1minut |
96oC 10 secunde |
50 oC 5 secunde |
60 oC 4 min |
4oC |
Purificarea produsilor de extensie - metoda de precipitare cu BigDye X-Terminator Purification Kit (AppliedBiosystems)
Peste produsii PCR vortexati se adauga solutie SAM.
Se adauga solutie XTerminator. Inainte de adaugarea solutiei XTerminator este necesara vortexarea acesteia cel putin 10 secunde sau pana cand solutia devine omogena.
Se vortexeaza tuburile 30 minute la 2000 rpm.
Se centrifugheaza tuburile timp de 2 minute la 2000 rpm.
Supernatantul se transfera in placa de lucru care se incarca in analizorul genetic automat ABI Prism 3130.
Secventele nucleotidice obtinute pentru regiunea mitocondriala D-loop au fost aliniate cu ajutorul programului BioEdit, corectate manual si comparate prin programul BLAST cu secventele existente in bancile de date.
Construirea arborilor filogenetici
Estimarea relatiilor filogenetice pornind de la informatia continuta in secventele ADN se bazeaza pe definirea acelei succesiuni de etape (algoritmi) prin care se poate construi cel mai bun arbore filogenetic. Un arbore filogenetic constituie reprezentarea grafica a filogeniei unui grup de organisme. Pentru obtinerea arborilor filogenetici se respecta urmatoarea succesiune de etape:
obtinerea secventelor ADN pentru analiza;
compararea secventelor obtinute cu cele din bancile de date;
alinierea secventelor;
selectarea metodelor filogenetice;
construirea arborilor si evaluarea acestora (Figura 37).
Alinierea secventelor
Baza fundamentala a unui arbore filogenetic este realizarea unui aliniament multiplu continand secventele de interes. Prin alinierea secventelor se confirma ca toate acestea sunt omoloage. Alinierea secventelor se poate realiza manual (folosind un editor text), automat (cu ajutorul unui software specializat) sau combinat.
Figura 37. Etapele unei analize filogenetice (D.W. Mount, Bioinformatics: Sequence and Genome Analysis, Cold Spring Harbor Press, 2004).
Secventele obtinute pentru regiunea D-loop la speciile de salmonide analizate au fost aliniate cu programul ClustalX. Pentru alinierea propriu-zisa a secventelor de interes cu programul ClustalX se realizeaza pasii urmatori:
se definesc parametrii pentru aliniere (gap penalty). O pereche de secvente poate fi aliniata astfel incat sa maximizam numarul de resturi nucleotidice care sunt comune, prin introducerea de spatii (gap) la una dintre secvente. Biologic, la nivelul acestor regiuni se presupune existenta unor insertii sau deletii care se produc atunci cand secventele diverg dintr-un ancestor comun. Este totusi necesar sa restrangem numarul de spatii introduse astfel incat aliniamentul sa aiba sens din punct de vedere biologic. De aceea este utilizat un sistem de notare astfel incat resturile nucleotidice verificate sa obtina un scor pozitiv si spatiile sa obtina un scor negativ sau gap penalty (penalizarea discontinuitatilor).
ClustalX creaza aliniamente multiple in trei etape: i) aliniaza individual fiecare secventa cu celelalte secvente intr-o serie de aliniamente perechi; ii) utilizeaza acest set de aliniamente perechi pentru a crea un arbore ghid; iii) utilizeaza acest arbore ghid pentru a crea un aliniament multiplu.
Alegerea metodelor de constructie a arborilor
In general se utilizeaza doua categorii de metode pentru construirea arborilor filogenetici:
metode de distanta (fenetice).
metode de caractere (cladistice).
Metodele de distanta (fenetice) propun construirea arborilor plecand de la asemanarile observate intre fiecare pereche de unitati evolutive si se bazeaza pe calculul unei matrice de distanta obtinuta comparand secventele doua cate doua si calculand numarul total de diferente pentru toate cuplurile posibile. Se construieste apoi un arbore filogenetic ale carui ramuri sunt cat mai apropiate de numerele continute de matricea de distanta. Cele mai utilizate metode de distanta sunt reprezentate de NJ (Neighbor Joing) si UPGMA (Unweighted Pair-Group Method with Arithmetic Means), iar acestea presupun construirea arborelui filogenetic prin succesiunea urmatoare de pasi: aliniere → matrice de distanta → arbore filogenetic.
Cea mai simpla metoda de a masura distanta dintre doua secvente este de a alinia secventele si de a numara diferentele dintre acestea. Gradul de divergenta se numeste distanta Hamming (d). Pentru un aliniament cu lungimea N si n situsuri, la nivelul caruia exista diferente d = n / N, distanta observata (D) dintre doua secvente este: D = 1 - S, unde S = M/L.
Similitudinea (S) dintre doua secvente reprezinta raportul dintre numarul de situsuri sinonime (M) si lungimea secventei (L). Distanta evolutiva dintre doua secvente este egala cu numarul de substitutii care sunt produse pe doua linii evolutive plecand de la un ancestor comun. Diferentele observate nu sunt egale cu distanta genetica, aceasta implicand mutatii care nu pot fi observate direct.
Convergenta, reversia si schimbarile multiple sunt cel mai adesea ascunse observatiei. Pentru a tine cont de ele, numeroase modele au fost dezvoltate pentru a corecta distantele observate. Astfel, Jukes si Cantor (1969) au propus o formula corectiva: D = -3/4 ln(1- 4/3p). Este un model simplu care presupune ca substitutiile sunt echiprobabile (α = β, unde α este probabilitatea de tranzitie, β este probabilitatea de transversie). In realitate, rata tranzitiilor este mai mare decat cea a transversiilor.
Modelul Kimura cu 2 parametrii (K2P) tine cont de proportia dintre numarul de tranzitii (α) si transversii (β). Tranzitiile sunt mult mai numeroase decat transversiile. Formula corectiva propusa este: dxy = -1/2 ln(1- 2P -Q) + 1/4 ln (1- 2Q), unde P = proportia de tranzitii si Q = proportia de transversii.
UPGMA (Unweighted Pair-Group Method with Arithmetic Means) este o metoda de clusterizare folosita in bioinformatica pentru crearea de arbori filogenetici printr-un algoritm de insumare secvential. Metoda se bazeaza pe regruparea secventelor celor mai apropiate din punct de vedere evolutiv. In matricea de distanta cele doua unitati taxonomice operationale, avand distanta cea mai mica, sunt adunate intr-un nou cluster compus, care va fi considerat in continuare ca o singura unitate operationala. Este creata o noua matrice, in care distantele celorlalte unitati taxonomice operationale ale clusterului sunt calculate. Procesul se repeta pana cand raman doar doua OTU. Acestea sunt insumate si radacina este amplasata la jumatatea distantei calculate dintre cele doua sume.
UPGMA este o metoda simpla, dezvoltata original pentru construirea fenogramelor taxonomice. Metoda se bazeaza pe asumarea unei rate constate a evolutiei sustinuta de ipoteza ceasului molecular si anume, distantele sunt ultrametrice si deci ele evolueaza cu o viteza constanta. Obtinerea de date ultrametrice este foarte improbabila, chiar daca rata substitutiei ar fi perfect constanta.
Metoda NJ (Neighbor Joing) este cea mai utilizata metoda de distanta atunci cand folosim criteriul minimei evolutii in constructia arborilor filogenetici. Ca si UPGMA, metoda NJ se bazeaza pe masurarea distantelor genetice pentru construirea unui arbore filogenetic, dar diferenta intre cele doua metode consta in faptul ca NJ tine cont de diferentele de viteza evolutiva intre diferitele ramuri ale arborelui filogenetic. Metoda NJ este mai eficienta decat celelate metode de distanta si utilizeaza un altgoritm polinominal la fel ca metodele de clusterizare. Metoda nu utilizeaza ipoteza orologiului molecular, algoritmul folosit in acest caz realizand calculul lungimii bratelor arborelui, astfel incat distantele deduse ale arborelui sa corespunda cu cele mai apropiate distante masurate intre secvente. Aceasta modalitate de calcul este ilustrata si de denumirea metodei Neighbor Joing - "cel mai apropiat vecin". Metoda genereaza arbori filogenetici fara radacina prin reunirea nodurilor care sunt apropiate unele de altele.
Metodele de distanta prezinta avantajul ca sunt metode rapide si permit analiza unui numar mare de date si testarea unui numar mare de ipoteze alternative. Dezavantajul acestora consta in pierderea unei parti de informatie la trecerea de la matricea bazata pe caractere la matricea bazata pe calculul distantelor. De asemenea, nu permit combinarea unor caractere diferite in aceeasi matrice (de ex. caractere morfologice si secvente ADN).
Metodele de caractere (cladistice)
Metoda MP (Maximum Parcimony) este o tehnica cladistica bazata pe parcimonie. Principiul parcimoniei postuleaza ca pentru un grup de specii, filogenia verosimila este cea care necesita cel mai mic numar de schimbari evolutive. Arborele filogenetic al speciilor este conceput implicand un numar minim de evenimente evolutive. Lungimea arborelui L este egala cu suma numarului de modificari I pentru fiecare situs k.
Etapele de analiza prin metoda MP sunt urmatoarele:
i. identificarea situsurilor informative;
ii.deducerea tuturor topologiilor arborilor posibili pentru secventele de date;
iii. calculul numarului minim de substitutii pentru fiecare situs informativ;
iv. calculul sumei de modificari pentru fiecare arbore;
v. alegerea topologiei arborelui care necesita cele mai putine modificari.
Metoda MP deduce arborele (sau arborii) cu cel mai mic numar de pasi necesari pentru a produce variatii ale secventei observate. MP prezinta o serie de avantaje: tine cont de tipurile de caractere, nu reduce informatia la un numar unic (o distanta), incerca sa furnizeze informatie despre secventele ancestrale si evalueaza arbori diferiti. Comparativ cu metodele de distanta este foarte lenta. De asemenea, prezinta dezavantajul de a nu utiliza decat partial informatia (situsuri informative), nu corecteaza substitutiile multiple si nu calculeaza lungimea bratelor.
Metoda ML (Maximum Likelyhood) sau "metoda probabilitatii maxime" este o alternativa la metoda MP. ML este o metoda statistica utilizata pentru a deduce parametri distributiei de probabilitate a unui anumit esantion. Din moment ce fiecare situs evolueaza independent, arborele este calculat separat pentru fiecare dintre acestia. Probabilitatea maxima este calculata pentru probabilitatea fiecarui rest dintr-un aliniament, pe baza unui model al procesului de substitutie. Prin metoda ML se incearca reconstruirea unei filogenii folosind un model explicit de evolutie. Metoda este cea mai eficienta cand este folosita pentru a testa sau a optimiza un arbore deja existent.
ML prezinta o serie de avantaje:
cea mai fiabila dintre metodele filogenetice;
ceea care conduce la rezultatul cel mai apropiat de arborele evolutiv real;
comparativ cu parcimonia, ea este mai consistenta si mai putin sensibila la efectele atractiei bratelor lungi.
permite aplicarea diferitelor modele evolutive (ex. modelul Kimura care tine cont de diferentele intre tranzitii si transversii) si estimarea lungimii bratelor in functie de schimbarea evolutiva.
Principalul dezavantaj este acela ca necesita o mare putere de calcul si mult timp, fiind cea mai lenta dintre toate metodele filogenetice.
Lucrarile publicate pe teme de filogenie propun folosirea unor algoritmi diferiti si a unor seturi de date diferite pentru obtinerea unor concluzii valabile. In unele cazuri metode diferite actioneaza sinergic. De exemplu, metoda NJ produce un singur arbore care este utilizat pentru a valida un arbore construit prin metodele MP sau ML. Folosind metode alternative putem sa obtinem indicatii despre robustetea concluziilor rezultate.
Construirea si evaluarea arborilor filogenetici
Cel mai adesea termenul de filogenie este identificat cu un arbore filogenetic. Acesta este o reprezentare grafica a filogeniei unui grup de organisme si este format din noduri si ramuri. Nodurile externe reprezinta unitati taxonomice operationale (OTU).
Exista mai multe tipuri de arbori (dendograme):
fenograma - o dendograma obtinuta prin metodele de distanta unde relatiile dintre taxoni exprima gradele de similitudine globala.
cladograma - o dendograma care exprima relatiile filogenetice dintre taxoni si construita plecand de la analiza cladistica.
filograma - o cladograma data de lungimea bratelor si proportionala cu numarul de schimbari evolutive.
Majoritatea metodelor filogenetice produc arbori fara radacina. Utilizarea unui outgroup (grup de comparatie), grup ales din exteriorul grupului studiat, duce la construirea unui arbore cu radacina. In cazul arborilor elaborati pe baza metodei fenetice, lungimea ramurilor reprezinta distanta genetica dintre taxoni, iar pentru cei elaborati prin metoda cladistica (cladograme) acestea se raporteaza la evenimentele evolutive (caractere derivate) care au avut loc pentru fiecare linie.
Testarea fiabilitatii arborilor se face prin analiza "bootstrap". Analiza "bootstrap" este o abordare folosita de obicei pentru a masura robustetea topologiei unui arbore. Analiza testeaza fiabilitatea ramurilor interne. Fiecare replicare "boostrap" produce o noua aliniere "artificiala"care este utilizata pentru a construi un arbore "artificial". Pentru fiecare ramura interna se calculeaza procentajul de arbori "artificiali" care contin aceasta ramificare.
In general, se considera ca ramurile definite printr-o valoare de incarcare mai mare 50% sunt fiabile. Sunt necesare 1000 de replicari de incarcare pentru ca aceasta metoda sa fie statistic valabila. Analiza "bootstrap" poate fi aplicata arborilor filogenetici construiti prin metodele Neighbor Joining, Maximum Parsimony si UPGMA. Fiecare set de date "bootstrap" este analizat din punct de vedere filogenetic.
Construirea arborilor filogenetici pe baza secventelor regiunii D-loop la salmonide a fost realizata prin metodele NJ si MP, utilizand programul PHYLIP (Phylogenetic Inference Package) v3.68.
REZULTATE SI DISCUTII
Optimizarea protocoalelor PCR pentru amplificarea regiunii mitocondriale D-loop
Initial s-a determinat temperatura de hibridizare optima pentru fiecare set de primeri prin PCR in gradient de temperatura. Ampliconii au fost verificati prin electroforeza in gel de agaroza 2%, obtinandu-se profile electroforetice din Figurile 38-41.
In final a fost optimizat protocolul de amplificare pentru regiunea mitocondriala D-loop la speciile de salmonide analizate si au fost alese temperaturile optime pentru hibridizarea primerilor.
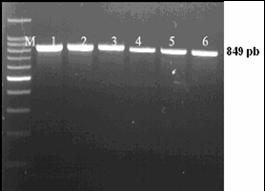
Figura 38. Electroforeza in gel de agaroza 2% pentru evidentierea produsilor reactiei PCR in gradient de temperatura pentru regiunea mitocondriala D-loop la Thymallus thymallus. M - Marker de masa moleculara 100 bp; 1-6 Fragmente obtinute la urmatoarele temperaturi de hibridizare: 1 - 59,1sC; 2 - 58,3sC; 3 - 55,9sC; 4 - 55,1sC; 5 - 54,4sC; 6 - 53,6 sC.
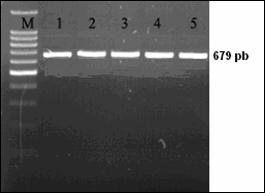
Figura 39.. Electroforeza in gel de agaroza 2% pentru evidentierea produsilor reactiei PCR in gradient de temperatura pentru regiunea mitocondriala D-loop la Salmo trutta fario. M - Marker de masa moleculara 100 bp; 1-5 Fragmente obtinute la urmatoarele temperaturi de hibridizare: 1 - 60sC; 2 - 59,1sC; 3 - 58,3sC; 4 - 57,4sC; 5- 56,6sC.
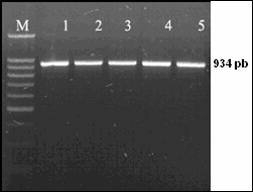
Figura 40. Electroforeza in gel de agaroza 2% pentru evidentierea produsilor reactiei PCR in gradient de temperatura pentru regiunea mitocondriala D-loop la Salvelinus fontinalis. M - Marker de masa moleculara 100 bp; 1-5 Fragmente obtinute la urmatoarele temperaturi de hibridizare: 1 - 55,9sC; 2 - 55,1sC; 3 - 54,4sC; 4 - 52,7sC; 5- 51,9sC.
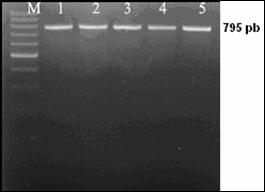
Figura 41. Electroforeza in gel de agaroza 2% pentru evidentierea produsilor reactiei PCR in gradient de temperatura pentru regiunea mitocondriala D-loop la Oncorhynchus mykiss. M - Marker de masa moleculara 100 bp; 1-5 Fragmente obtinute la urmatoarele temperaturi de hibridizare: 1 - 51sC; 2 - 52,7sC; 3 - 55,1sC; 4 - 57,4sC; 5- 59,1sC.
Optimizarea protocoalelor PCR pentru amplificarea unei regiuni extinse din genomul mitocondrial in vederea realizarii tehnicii PCR-RFLP
Tehnica PCR-RFLP se bazeaza pe compararea profilelor de restrictie rezultate in urma digestiei ADN obtinut prin amplificare cu endonucleaze de restrictie. Eventualele mutatii care apar la nivelul secventei ADN pot crea sau modifica un situs de restrictie avand drept rezultat obtinerea de fragmente de restrictie diferite.
Enzimele de restrictie au fost descoperite in urma studierii fenomenului de rezistenta bacteriana la atacul bacteriofagilor, determinat de sinteza unor enzime de restrictie cu rol in degradarea materialului genetic fagic si protejarea celui propriu.
Descoperirea, purificarea si caracterizarea unui numar foarte mare de astfel de enzime a impus adoptarea unei nomenclaturi universale. Astfel, pentru denumirea lor se foloseste un cod de trei litere, in format italic, prima litera semnificand genul, iar celelalte doua specia bacteriana de la care a fost izolata enzima. (ex: Eco - Escherichia coli). In unele cazuri, o a patra litera, in format normal, va indica tulpina bacteriana. Daca o anumita tulpina bacteriana are mai mult de un sistem de restrictie-modificare, acestea se vor identifica prin numere romane.
Endonucleazele de restrictie sunt enzime care recunosc secvente de ADN specifice, scurte, recunoasterea fiind urmata de legare si apoi clivare, fie la nivelul situsului, fie la o distanta oarecare de acesta. Sistemele de restrictie-modificare (pot indeplini si functia de metilare) au fost impartite in trei categorii: I, II si III. Categoriile I si III constau in enzime care au atat functii de restrictie cat si functii de metilare. Ambele tipuri recunosc secvente specifice, nemetilate, de ADN dublu catenar. Enzimele din categoria I cliveaza ADN intr-o maniera situs-nespecifica, iar cele din categoria III taie la nivelul unor situsuri specifice situate la o distanta de 25-27 nucleotide de secventa de recunoastere. In categoria II sunt incluse toate enzimele cu capacitate de restrictie-modificare care actioneaza exact la nivelul situsurilor de recunoastere pe care le scindeaza sau modifica.
Experimental, se recomanda ca pentru o anumita secventa ADN sa se identifice posibilele situsuri de restrictie caracteristice genotipului normal. Ulterior se verifica daca mutatiile pe care dorim sa le identificam afecteaza vreunul dintre aceste situsuri. Acestea pot modifica un situs de restrictie deja existent sau pot genera un situs nou. In abordarea moderna, tehnica RFLP se realizeaza prin amplificarea zonei de interes prin reactie PCR si digestia cu enzime de restrictie a ampliconilor obtinuti. Ea poate fi folosita pentru genotipare individuala, identificarea de polimorfisme normale si patologice si identificarea profilelor de restrictie ale microorganismelor.
In final a fost optimizat protocolul de amplificare pentru regiunea mitocondriala extinsa la speciile de salmonide analizate si au fost alese temperaturile optime pentru hibridizarea primerilor.
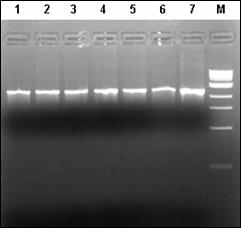
Figura 42.. Electroforeza in gel de agaroza 2% pentru evidentierea produsilor reactiei PCR in gradient de temperatura pentru regiunea mitocondriala extinsa la Oncorhynchus mykiss. M - Marker de masa moleculara 1 Kb; 1-7 Fragmente obtinute la urmatoarele temperaturi de hibridizare: 1 - 51sC; 2 - 51,9sC; 3 - 53,3sC; 4 - 55,2sC; 5 - 58,1sC; 6 - 60,1sC; 7 - 61,4sC.
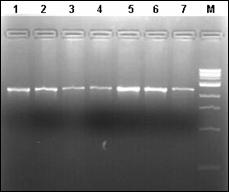
Figura 43.. Electroforeza in gel de agaroza 2% pentru evidentierea produsilor reactiei PCR in gradient de temperatura pentru regiunea mitocondriala extinsa la Thymallus thymallus. M - Marker de masa moleculara 1 Kb; 1-7 Fragmente obtinute la urmatoarele temperaturi de hibridizare: 1 - 51sC; 2 - 51,9sC; 3 - 53,3sC; 4 - 55,2sC; 5 - 58,1sC; 6 - 60,1sC; 7 - 61,4sC.
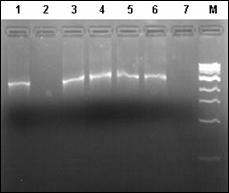
Figura 44.. Electroforeza in gel de agaroza 2% pentru evidentierea produsilor reactiei PCR in gradient de temperatura pentru regiunea mitocondriala extinsa la Hucho hucho M - Marker de masa moleculara 1 Kb; 1-7 Fragmente obtinute la urmatoarele temperaturi de hibridizare: 1 - 51sC; 2 - 51,9sC; 3 - 53,3sC; 4 - 55,2sC; 5 - 58,1sC; 6 - 60,1sC; 7 - 61,4sC.
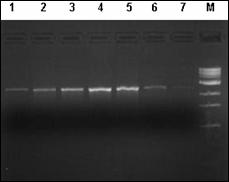
Figura 45.. Electroforeza in gel de agaroza 2% pentru evidentierea produsilor reactiei PCR in gradient de temperatura pentru regiunea mitocondriala extinsa la Salmo trutta labrax M - Marker de masa moleculara 1 Kb; 1-7 Fragmente obtinute la urmatoarele temperaturi de hibridizare: 1 - 51sC; 2 - 51,9sC; 3 - 53,3sC; 4 - 55,2sC; 5 - 58,1sC; 6 - 60,1sC; 7 - 61,4sC.
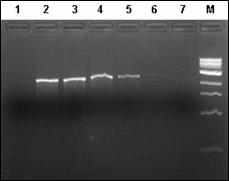
Figura 46.. Electroforeza in gel de agaroza 2% pentru evidentierea produsilor reactiei PCR in gradient de temperatura pentru regiunea mitocondriala extinsa la Salvelinus fontinalis M - Marker de masa moleculara 1 Kb; 1-7 Fragmente obtinute la urmatoarele temperaturi de hibridizare: 1 - 51sC; 2 - 51,9sC; 3 - 53,3sC; 4 - 55,2sC; 5 - 58,1sC; 6 - 60,1sC; 7 - 61,4sC.
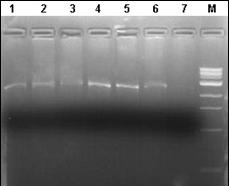
Figura 47.. Electroforeza in gel de agaroza 2% pentru evidentierea produsilor reactiei PCR in gradient de temperatura pentru regiunea mitocondriala extinsa la Salmo trutta M - Marker de masa moleculara 1 Kb; 1-7 Fragmente obtinute la urmatoarele temperaturi de hibridizare: 1 - 51sC; 2 - 51,9sC; 3 - 53,3sC; 4 - 55,2sC; 5 - 58,1sC; 6 - 60,1sC; 7 - 61,4sC.
Analiza produsilor de restrictie ai regiunii mitocondriale extinse de la salmonide
Scopul nostru a fost implementarea unei metode simple si eficiente care sa fie utilizata pentru a detecta cu precizie exemplarele apartinand diferitelor specii de salmonide din Romania. Identificarea s-a realizat prin amplificarea cu ajutorul tehnicii PCR a unei regiuni mitocondriale extinse (care cuprinde regiunea D-loop, dar si gene codoficatoare pentru ARNr si ARNt) cu primeri specifici, urmata de digestia ampliconilor cu enzima de restrictie Hinf I (Figurile 48-49).
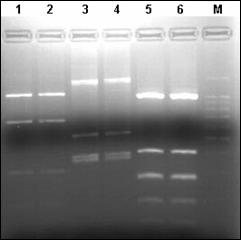
Figura 48. Electroforeza in gel de agaroza 2,5% pentru evidentierea produsilor reactiei de restrictie cu Hinf I pentru regiunea mitocondriala extinsa la 3 specii de salmonide. M - Marker de masa moleculara 100 pb; 1, 2 - Salvelinus fontinalis, 3, 4 - Hucho hucho, 5, 6 - Thymallus thymallus.
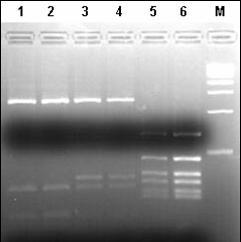
Figura 49. Electroforeza in gel de agaroza 2,5% pentru evidentierea produsilor reactiei de restrictie cu Hinf I pentru regiunea mitocondriala extinsa la 3 specii de salmonide. M - Marker de masa moleculara 100 pb; 1, 2 - Salmo trutta labrax, 3, 4 - Salmo trutta, 5, 6 - Onchorhyncus mykiss.
In urma analizei Figurilor 48 si 49 se observa ca scindarea ampliconilor cu endonucleaza de restrictie Hinf I, urmata de analiza produsilor rezultati in gel de agaroza poate identifica diferentele dintre diferitele specii de salmonide. Tehnica pusa astfel la punct poate fi utilizata cu succes in identificarea moleculara a diferitelor specii de salmonide si poate fi testata si pentru analize moleculare ale hibrizilor dintre diferite specii. Detectarea acestor hibrizi in mediul natural ar reprezenta o realizare majora in sustinerea eforturilor de conservare a fondului salmonicol.
Analiza regiunii mitocondriale D-loop si construirea arborilor moleculari
Fragmentele obtinute pentru regiunea de control mitocondriala (D-loop) la speciile de salmonide din Romania au fost secventiate si comparate cu secvente similare din baza de date GenBank. Initial, secventele au fost analizate si corectate manual cu ajutorul programului BioEdit. Alinierea secventelor a fost realizata cu programul ClustalX.
Pentru reprezentantii speciei Salmo trutta fario (pastrav indigen), secventa pentru regiunea D-loop a fost aliniata cu secvente similare din GenBank. Secventele din baza de date reprezinta haplotipuri mitocondriale diferite si provin de la indivizi de pastrav comun cu distributie geografica heterogena. Astfel, au fost selectate din baza GenBank haplotipuri apartinand liniilor evolutive: Atlantica (Nord Atlantica/Sud Atlantica), Adriatica, Mediteraneana, Danubiana si Duero (Tabel 11). Pentru Salmo marmoratus nu am identificat in GenBank decat secvente de lungime mica si astfel, haplotipurile apartinand reprezentantilor de Salmo marmoratus nu au fost introduse in analiza.
Tabel 11. Secvente din GenBank folosite in aliniamentul cu programul ClustalX.
|
Specie |
Numar de acces in GenBank |
Observatii |
|
Salmo trutta |
AF253544 |
Haplotip Duero |
|
AF253545 |
Haplotip Duero |
|
|
AF253546 |
Haplotip Nord Atlantic |
|
|
AF253547 |
Haplotip Mediteranean |
|
|
AF253549 |
Haplotip Mediteranean |
|
|
AF253550 |
Haplotip Mediteranean |
|
|
AF253551 |
Haplotip Adriatic |
|
|
AF253552 |
Haplotip Adriatic |
|
|
AF253553 |
Haplotip Sud Atlantic |
|
|
AF253555 |
Haplotip Sud Atlantic |
|
|
AF253556 |
Haplotip Sud Atlantic |
|
|
AF253557 |
Haplotip Sud Atlantic |
|
|
AF253558 |
Haplotip Nord Atlantic |
|
|
GQ284832 |
Haplotip Danubian |
|
|
GQ284833 |
Haplotip Danubian |
|
|
GQ284834 |
Haplotip Danubian |
|
|
GQ284835 |
Haplotip Danubian |
|
|
GQ284836 |
Haplotip Danubian |
|
|
GQ284837 |
Haplotip Danubian |
|
|
NC_010007 |
Salmo trutta trutta (pastrav de mare) |
|
|
X93586 |
Tara de provenienta - Spania |
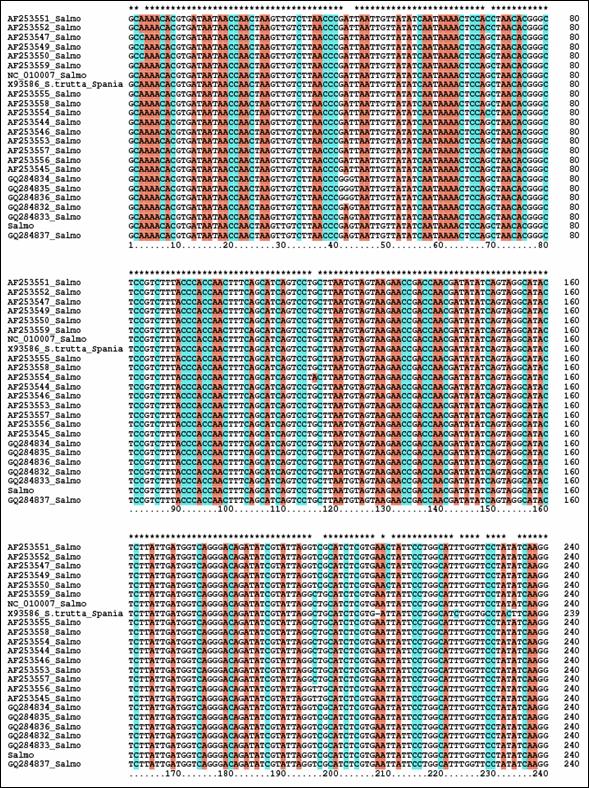
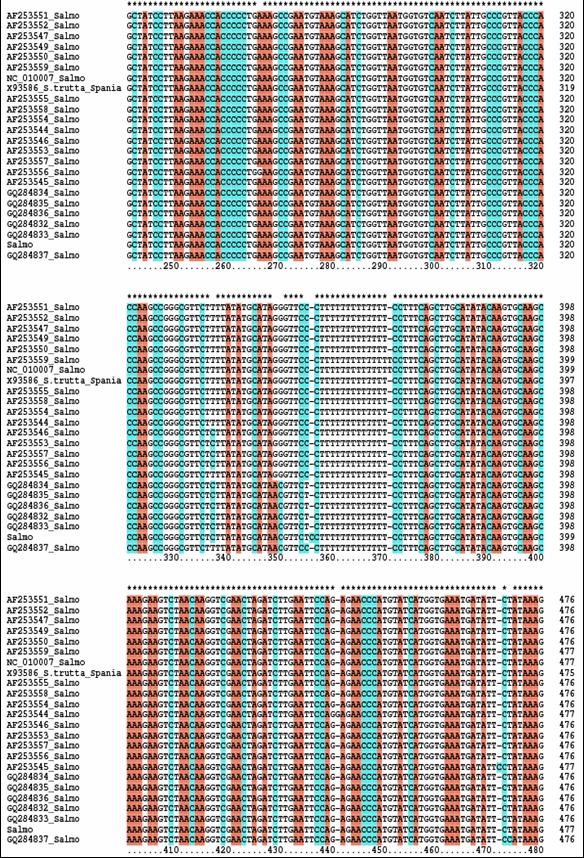
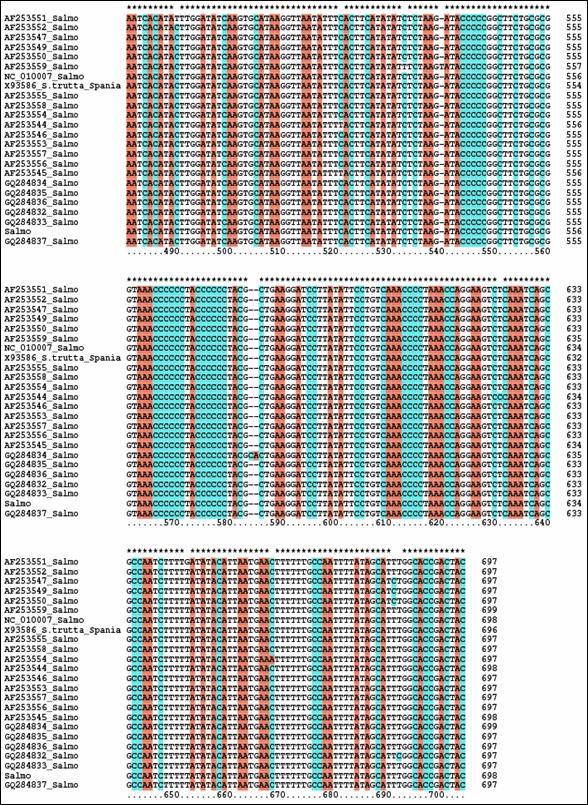
![]()
![]()
![]()
![]()
![]() Figura
50. Aliniere folosind
programul ClustalX a secventelor pentru regiunea D-loop reprezentand
diverse haplotipuri.
Figura
50. Aliniere folosind
programul ClustalX a secventelor pentru regiunea D-loop reprezentand
diverse haplotipuri.
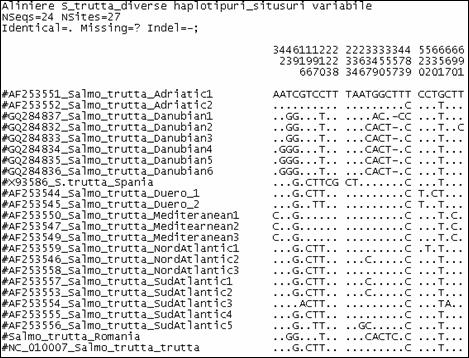
Figura 51. Situsurile variabile din fragmentul D-loop de la Salmo trutta. Numerele reprezinta pozitia ocupata in alinimaentul realizat cu ClustalW. Situsurile identice sunt indicate prin simbolul " ∙ ", iar gap-urile (deletiile) sunt indicate prin simbolul "-".
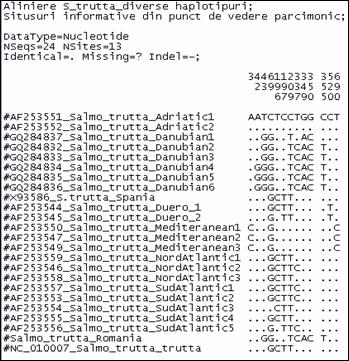
Figura 52. Situsurile informative din punct de vedere parcimonic din fragmentul D-loop de la Salmo trutta. Numerele reprezinta pozitia ocupata in aliniamentul realizat cu ClustalW. Situsurile identice sunt indicate prin simbolul " ∙ ", iar gap-urile (deletiile) sunt indicate prin simbolul "-".
Pentru Salmo trutta din Romania a fost identificat un singur haplotip pentru fragmentul din regiunea de control mitocondriala D-loop analizat si acesta a fost aliniat cu alte 23 de secvente pentru acelasi marker mitocondrial din GenBank (Figura 50). Din cele 703 situsuri nucleotidice (inclusiv situsurile gap) ale fragmentului analizat, 671 sunt situsuri conservate, iar 27 sunt situsuri variabile (Figura 51). Dintre cele 27 de situsuri variabile, 13 sunt situsuri informative din punct de vedere parcimonic (Figura 52), iar 14 reprezinta variatii de tip Singleton. In ceea ce priveste polimorfismele identificate la nivelul celor 24 de secvente analizate, numarul tranzitiilor (inlocuirea unei purine/pirimidine cu o baza de acelasi tip) este mai mare decat numarul transversiilor (inlocuirea unei purine cu o pirimidina si viceversa), raportul tranzitii/transversii (si/sv) fiind egal cu 2,5. Polimorfismele identificate la nivelul regiunii D-loop analizate sunt mutatii cu frecventa aleatoare si cu rol evolutiv.
In cazul speciei Oncorhynchus mykiss, probele biologice au provenit din diferite pastravarii din Romania. Dintre toate speciile de salmonide din tara noastra, pastravul curcubeu se preteaza cel mai bine la cresterea intensiva. In Romania aceasta, specie se creste exclusiv in pastravarii pentru consum alimentar.
In cazul probelor de pastrav curcubeu analizate de noi, secventele nucleotidice obtinute pentru un fragment din regiunea de control D-loop au fost aliniate cu secvente similare din GenBank reprezentand haplotipuri diferite cu ajutorul programului Clustal X (Figura 53). In cazul secventelor provenind de la Onchorhyncus mykiss din Romania au fost identificate trei haplotipuri diferite pentru regiunea D-loop. In cazul celor cinci secvente analizate (trei reprezentand secvente pentru reprezentantii din Romania si doua din GenBank) au fost identificate mai multe polimorfisme de tipul mutatiilor punctiforme - tranzitii, transversii, deletii (Tabel 12).
Tabel 12. Polimorfisme identificate in secventa nucleotidica a fragmentului D-loop la specia Oncorhynchus mykiss. Numerele reprezinta pozitia ocupata raportat la haplotipul Romania 3.
|
Specia |
Haplotip |
Polimorfism |
||||
|
476 | ||||||
|
Onchorhyncus mykiss |
Haplotip Romania1 |
A |
C |
Deletie T |
C |
C |
|
Haplotip Romania2 |
T |
T |
T |
T |
T |
|
|
Haplotip Romania3 |
A |
C |
T |
T |
T |
|

Figura 53. Aliniere folosind programul ClustalX a secventelor pentru regiunea D-loop de la specia Oncorhynchus mykiss din Romania cu secventa similara din GenBank (L29771 si S68946).
La Salvelinus fontinalis, au fost secventiate si comparate probe ADN provenind de la indivizi din bazine hidrografice distincte: raul Bratia (bazin Arges), raul Gilau (bazin Lotru) si ferma RNP Bradisor (judetul Valcea) si a fost identificat un singur haplotip mitocondrial.
Secventa obtinuta pentru un fragment din regiunea de control mitocondriala D-loop a fost aliniata si comparata cu o secventa similara din GenBank (AF154850) (Figura 54). Aliniamentul realizat cu programul ClustalX arata ca intre secventele nucleotidice provenid de la indivizii de pastrav fantanel din Romania si o secventa omoloaga din GenBank exista un numar relativ crescut de polimorfisme (Tabel 13).
![]()
Figura 54. Aliniere folosind programul ClustalX a secventelor pentru regiunea D-loop de la specia Salvelinus fontinalis din Romania cu secventa similara din GenBank (AF154850).
Tabel 13. Polimorfisme identificate in secventa nucleotidica a fragmentului D-loop la specia Salvelinus fontinalis. Numerele reprezinta pozitia ocupata raportat la haplotipul Romania.
|
Specia |
Haplotip |
Polimorfism |
|||||||
|
Salvelinus fontinalis |
Romania |
delT |
A |
delA |
delT |
delT |
delC |
delA |
delT |
|
AF154850 |
T |
delA |
A |
T |
T |
C |
A |
T |
|
|
Romania | |||||||||
|
delT |
delA |
T |
T |
T |
A |
T | |||
|
AF154850 |
T |
A |
delT |
delT |
delT |
delA |
delT | ||
In cazul probelor de lipan analizate de noi, secventa obtinuta pentru un fragment din regiunea mitocondriala D-loop a fost aliniata cu diferite haplotipuri din baza de date GenBank cu ajutorul programului Clustal X (Figura 55). Au fost identificate un numar de 23 de situsuri variabile la nivelul secventelor analizate (Tabel 14), tranzitiile fiind mai numeroase decat transversiile, raportul si/sv fiind de 1,8.

Figura 55. Aliniere folosind programul ClustalX a secventelor pentru regiunea D-loop de la specia Thymallus thymallus din Romania cu secventa similara din GenBank (AF154850).
Tabel 14. Polimorfisme identificate in secventa nucleotidica a fragmentului D-loop la specia Thymallus thymallus. Numerele reprezinta pozitia ocupata raportat la haplotipul Romania.
|
Specia |
Haplotip |
Polimorfism |
|||||||
|
Thymallus thymallus |
Romania |
C |
C |
T |
A |
A |
T |
G |
G |
|
FJ852655 |
T |
T |
C |
G |
G |
C |
T |
delG |
|
|
AY841360 |
C |
C |
T |
A |
A |
T |
G |
delG |
|
|
AF522430 |
T |
T |
T |
A |
A |
T |
T |
delG |
|
|
Romania | |||||||||
|
C |
T |
G |
G |
A |
A |
T |
T |
||
|
FJ852655 |
delC |
C |
A |
G |
C |
C |
A |
A |
|
|
AY841360 |
C |
T |
G |
A |
C |
T |
T |
T |
|
|
AF522430 |
delC |
C |
A |
G |
C |
C |
A |
A |
|
|
Romania | |||||||||
|
C |
C |
G |
G |
delA |
G |
G | |||
|
FJ852655 |
G |
T |
T |
A |
A |
delG |
delG | ||
|
AY841360 |
C |
C |
G |
A |
A |
delG |
delG | ||
|
AF522430 |
G |
T |
T |
A |
A |
delG |
delG | ||
Secventele obtinute pentru regiunea D-loop au fost utilizate pentru stabilirea relatiilor filogenetice la speciile de salmonide cu ajutorul programului MEGA 4.0.
Regiunea de control mitocondriala este considerata un marker filogenetic si filogeografic foarte important atat la salmonide, cat si la pesti in general. La salmonide, studiile anterioare pe baza acestui marker molecular, au stabilit existenta a cinci grupuri filogeografice: atlantic, mediteranean, adriatic, danubian si marmoratus (Bernatchez et al., 1992; Osinov & Bernatchez, 1996; Bernatchez, 2001). Aria de distributie a grupului atlantic este foarte vasta, de la Muntii Atlas din Africa de Nord, pe coasta atlantica a Europei, si pana la Marea Barents. Bazinele Marilor Neagra, Caspica si Aral, dar si afluentii tributari acestora sunt populate de populatii diferite de Salmo trutta apartinand grupului danubian (Bernatchez & Osinov, 1995; Osinov & Bernatchez, 1996). Conform analizei secventelor nucleotidice nucleare pentru Internal Transcribed Spacer, populatiile de pastrav comun apartinand acestor trei bazine sunt cele mai vechi din punct de vedere filogenetic (Antunes et al., 2002; Presa et al., 2002).
Pe baza secventelor D-loop de la Salmo trutta au fost construiti arbori filogenetici utilizand metodele Neibourgh Joining si Minimum Evolution implementate in programul filogenetic MEGA 4.0. Distantele evolutive au fost stabilite cu ajutorul aplicatiei Modeltest (Posada & Crandall, 1998) si a fost selectata metoda Tamura 3-Parameter plus Gamma (Tammura, 1992). Rata de variatie intre situsurile nucleotidice a fost modelata cu o distributie "Gamma", parametrul α fiind egal cu 0,04. Arborii rezultati au fost reprezentati sub forma unor cladograme rectangulare (Figurile 56-57).
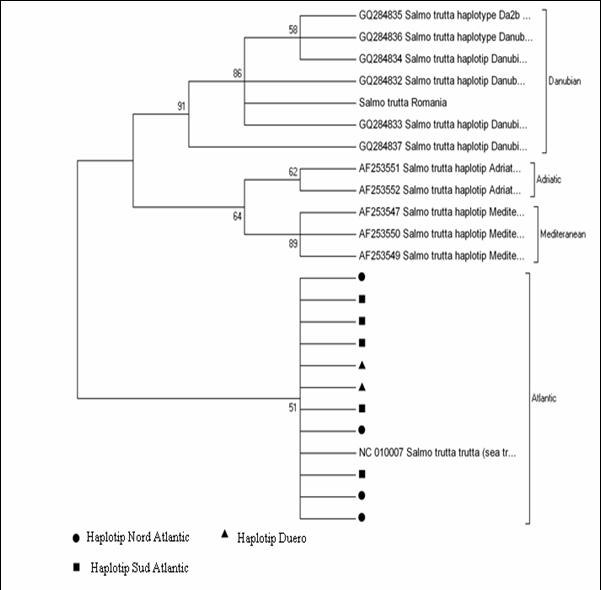
Figura 56. Reprezentarea relatiilor filogenetice existente intre diferite populatii de Salmo trutta stabilite pe baza regiunii mitocondriale D-loop. Metoda NJ. Valoare de bootstrap-1000 replicari.
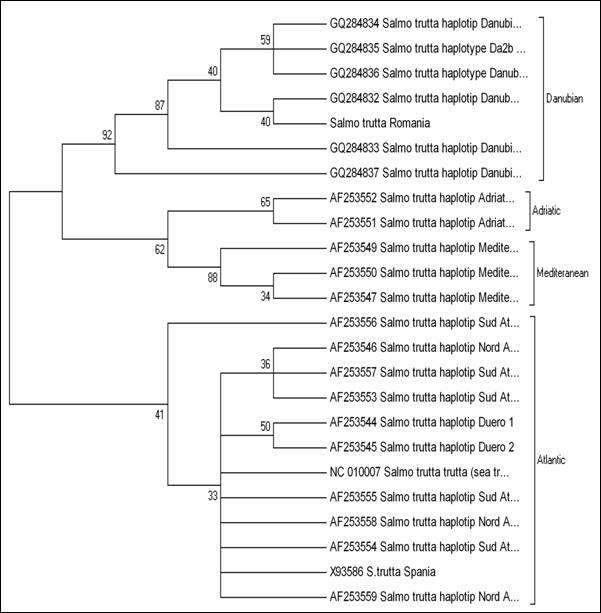
Figura 57. Reprezentarea relatiilor filogenetice existente intre diferite populatii de Salmo trutta stabilite pe baza regiunii mitocondriale D-loop. Metoda Minimum Evolution (ME). Valoare de bootstrap-1000 replicari.
Prin ambele metode au fost evidentiate doua clade distincte. Prima clada este constituita din haplotipuri de tipul nord-atlantic, sud-atlantic si Duero. Cea de-a doua clada grupeaza liniile evolutive adriatica, mediteraneana si danubiana. Rezultatele noastre confirma unele studii anterioare (Bernatchez et al., 2001). Cea mai veche separare a liniilor evolutive in cazul Salmo trutta a implicat fragmentarea allopatrica in trei mari subdivizuni corespunzatoare bazinelor Atlantic (linia atlantica), Ponto-Caspic (linia danubiana) si Mediteranean, urmata de o fragmentare ulterioara in interiorul bazinului Mediteranei, ceea ce a condus la divergenta liniilor mediteraneana, adriatica si marmoratus.
O prima validare a rezultatelor noastre este gruparea haplotipului din Romania in grupul danubian, ceea ce dovedeste ca in tara noastra exista inca populatii native de pastrav comun si ca repopularea bazinelor hidrografice cu indivizi reprodusi artificial s-a facut pornind de la genitori apartinand grupului danubian.
Haplotipurile nord-atlantic, sud-atlantic si Duero formeaza un grup monofiletic si sunt constituite in clada atlantica. Distributia haplotipurilor Duero impreuna cu cele nord-atlantice si sud-atlantice in cadrul acestei clade nu este deloc surprinzatoare.
Indivizii cu haplotip Duero provin din bazinul hidrografic cu acelasi nume din Peninsula Iberica. Studiile anterioare au confirmat rolul important al acestei zone, de refugiu al speciilor de salmonide in timpul ultimei glaciatiuni si in recolonizarea postglaciara. In interiorul bazinului fluviului Duero au fost identificate pana acum doua grupuri de haplotipuri: unul dintre acesta este inrudit cu linia atlantica (caracteristica populatiilor de pastrav comun din nord-estul Europei), celalalt grup este caracteristic regiunii interioare a acestui bazin. De asemenea, exista date care sugereaza existenta unui fenomen de hibridizare intre indivizii apartinand populatiei atlantice si celei Duero in interiorul acestui fluviu (Bouza et al., 2007).
Pentru stabilirea relatiilor filogenetice in cadrul familiei Salmonidae, pe langa secventele obtinute de noi pentru regiunea de control mitocondriala D-loop, am utilizat secvente similare din baza de date GenBank de la alte specii de salmonide in scopul realizarii unei analize filogenetice mai complexe (Tabel 15).
Tabel 15. Secvente din GenBank utilizate in analiza filogenetica la salmonide.
|
Specia |
Numar de acces (GenBank) |
|
Brachymystax lenok |
EU760491 |
|
Coregonus lavaraetus |
NC_002646 |
|
Hucho hucho |
EU729361 |
|
Hucho taimen |
EU760489 |
|
Oncorhynchus clarkii |
AY886762 |
|
Oncorhynchus keta |
NC_009261 |
|
Oncorhynchus kisutch |
NC_009263 |
|
Oncorhynchus masou |
NC_008747 |
|
Oncorhynchus mykiss |
L29771 |
|
Oncorhynchus nerka |
NC_008615 |
|
Oncorhynchus tshawytscha |
NC_009263 |
|
Plecoglossus altivelis |
NC_002734 |
|
Salmo trutta (haplotip Nord Atlantic) |
AF253559 |
|
Salmo trutta (haplotip Sud Atlantic) |
AF253557 |
|
Salmo trutta (haplotip Adriatic) |
AF253552 |
|
Salmo trutta (haplotip Duero) |
AF253544 |
|
Salmo trutta (haplotip Danubian) |
GQ284837 |
|
Salmo salar |
AF133701 |
|
Salvelinus alpinus |
NC_000861 |
|
Thymallus thymallus |
DQ439975 |
|
Thymallus articus |
NC_002734 |
Prin metodele NJ si Maximum Parcimony (MP) au fost construiti arbori care ilustreaza relatiile filogenetice intre cele 17 specii de salmonide analizate (Figurile 58-59). Specia Plecoglossus altivelis care apartine familiei Osmeridae a fost aleasa ca outgroup. Arborii obtinuti prin cele doua metode prezinta o topologie similara, singura diferenta fiind dispunerea genului Salvelinus in raport cu genurile Oncorhynchus si Salmo.
In cazul metodei NJ, modelul de distanta folosit a fost Tamura-Nei plus Gamma stabilit in Modeltest (Posada & Crandall, 1998). Rata de variatie intre situsurile nucleotidice a fost modelata cu o distributie "Gamma", parametrul α fiind egal cu 0,63. Analiza filogenetica a evidentiat trei mari clade corespunzatoare celor trei subfamilii: Thymallinae, Coregoninae si Salmoninae.
La baza arborelui filogenetic sunt dispuse speciile primitive de salmonide, cum ar fi Thymallus thymallus, Thymallus articus si Coregonus lavaraetus, apartinand subafmiliilor Thymallinae si Coregoninae, subafamilii care au aparut inaintea subfamiliei Salmoninae. Subfamilia Salmoninae este alcatuita din genurile Salmo, Oncorhynchus, Hucho, Brachymistax si Salvelinus, iar, conform analizei noastre, acestea formeaza grupuri monofiletice. Dintre Salmoninae, cele mai primitive specii sunt cele apartinand genurilor Brachymistax si Hucho.
Reprezentantii genului Salmo formeaza o clada distincta, in care Salmo salar sta la originea celorlalte specii. Haplotipurile nord-atlantic, sud-atlantic si Duero formeaza un grup monofiletic inrudit cu cel format de haplotipurile adriatic si mediteranean. Haplotipul din Romania si secventa introdusa in GenBank ca apartinand liniei evolutive danubiene formeaza grupuri monofiletice distincte.
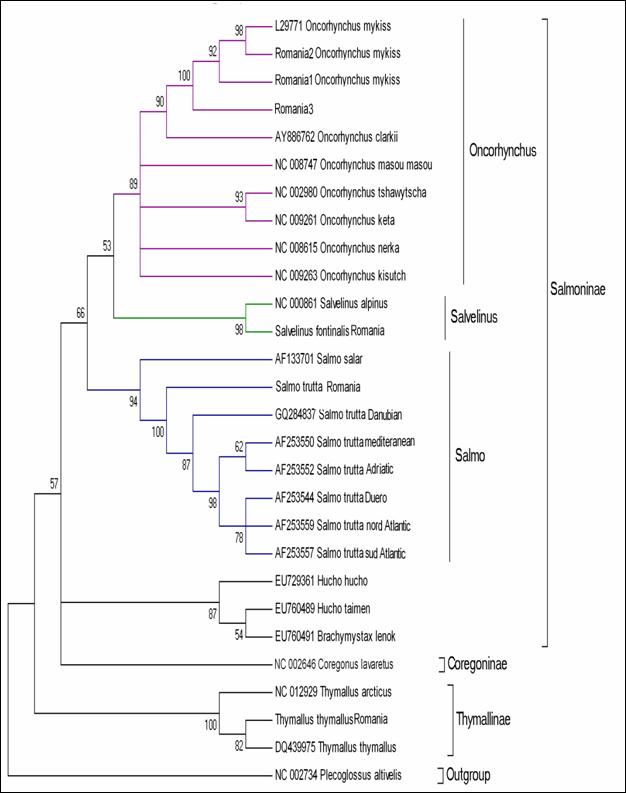
Figura 58. Arbore filogenetic construit prin metoda NJ (model Tamura-Nei + gamma). Valoare de bootstrap-1000 replicari.
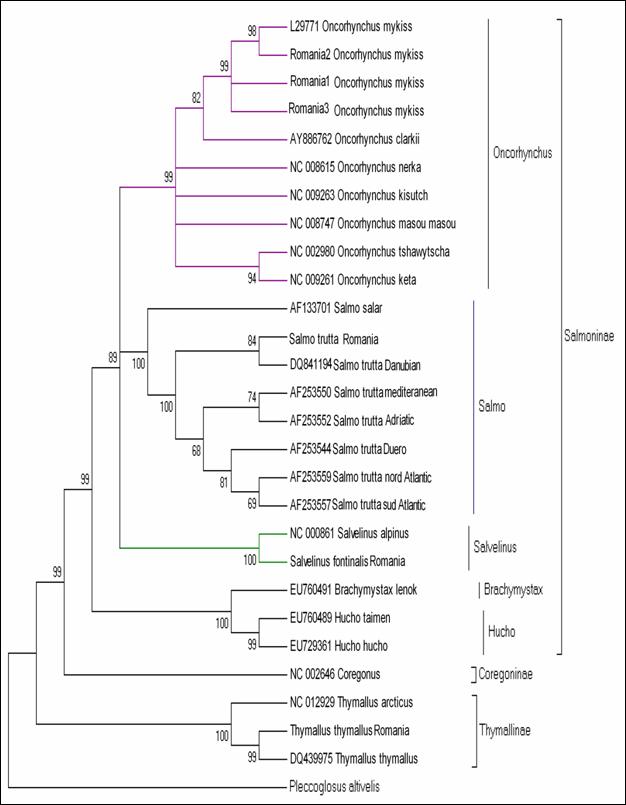
Figura 59. Arbore filogenetic construit prin metoda MP. Valoare de bootstrap-1000 replicari.
Speciile
genului Oncorhynchus formeaza o
clada distincta fata de celelalte grupuri de salmonide. Haplotipurile
din Romania se incadreaza in grupul celorlalte haplotipuri de la O. mykiss. Speciile O. mykiss si O. clarkii
formeaza un grup monofiletic, acestea fiind specii din Oceanul Pacific
care constituie o linie filogenetica distincta, si anume linia
"pastravului de Pacific" (Pacific Trout Lineage).
Speciile O. tshawytscha si O. keta formeaza un grup monofiletic, constituind una din liniile evolutive ale grupului somonului de Pacific (Pacific Salmon Group). Speciile O. masou masou, O. nerka si O. kisutch constituie grupuri monofiletice distincte si impreuna formeaza cea de-a doua linie evolutiva a grupului somonului de Pacific.
Speciile genului Salvelnius (Salvelinus fontinalis si Salvelinus alpinus) formeaza un grup monofiletic plasat in cadrul Salmoninaelor intre speciile genului Salmo si Onchorhynchus. Pozitia ocupata de Salvelinus contrazice ipoteza filogeniei clasice conform careia genurile Salmo si Oncorhynchus sunt cel mai apropiate din punct de vedere filogenetic si vin in sprijinul anumitor date de filogenie moleculara care afirma ca intre genurile Salvelinus si Oncorhynchus exista o apropiere filogenetica mai mare. Oakley & Phillips (1999), au stabilit relatiile filogenetice la 18 specii de salmonide utilizand un marker molecular nuclear (intronii genelor GH1 si GH2) si au ridicat ipoteza ca Salmo si Oncorhynchus nu sunt grupuri taxonomice direct inrudite.
Crespi et al., (2003) au realizat un studiu complex de filogenie la 30 de specii de salmonide, utilizand ca markeri filogenetici 16 gene mitocondriale si 9 gene nucleare. Printre concluziile acestui studiu se numara si faptul ca (a) Salmo reprezinta un grup inrudit cu grupul (Oncorhynchus, Salvelnius), (b) Salvelinus este taxonul cel mai apropiat din punct de vedere filogenetic de Oncorhynchus. Concluziile anterioare au fost bine sustinute de valorile de bootstrap obtinute si de analiza de tip bayesian. Inrudirea intre Salvelinus si Oncorhynchus a fost sustinuta de analiza genelor GH1C, VIT si ND3 si de datele nucleare combinate.
In cazul analizei noastre bazate pe secventa D-loop prin NJ sunt sustinute datele moleculare mentionate anterior. In schimb, metoda MP confirma ipoteza filogeniei clasice conform careia Salmo si Oncorhynchus reprezinta taxoni foarte apropiati din punct de vedere evolutiv.
BIBLIOGRAFIE
Allendorf, F.W., Thorgaard, G.H. 1984. Tetraploidy and the evolution of salmonid fishes. In ''Evolutionary Genetics of Fishes'' (B. J. Turner, Ed.), pp.1-53, New York.
Alves-Gomes, J.A., Ortı´, G., Haygood M., Heiligenberg W., 1995. Phylogenetic Analysis of the South American Electric Fishes (Order Gymnotiformes) and the Evolution of Their Electrogenic System: A Synthesis Based on Morphology, Electrophysiology, and Mitochondrial Sequence Data. Molecular Biology and Evolution 12, 2, pp. 298-318.
Apostolidis A. P., Loukovitis D., Tsigenopoulos C. S. (2007) Genetic characterization of brown trout (Salmo trutta) populations from the Southern Balkans using mtDNA sequencing and RFLP analysis. Hydrobiologia, Springer.
Anderson S, Bankier A.T, Barrell B.G, De Bruijn M.H., Coulson A.R., Drouin J, Nierlich D.P., Roe B.A., Sanger F, Schreier P.H., Smith AJ, Staden R., Young I.R. 1981. Sequence and organization of the human mitochondrial genome. Nature 290: 457-465.
Antipa, G. 1909. Fauna Ichtiologica a Romaniei. Inst. Arte Grafice Carol Göbl, Bucuresti: 200-209.
Aquadro, C.F. and Greenberg, B. 1983. Human mitochondrial DNA variation and evolution: analysis of nucleotide sequences from seven individuals. Genetics, 103, 287-312.
Banarescu, P. (1964) Fauna Republicii Populare Romine. Piscies-Osteichthyes (Pesti ganoizi si ososi) Volumul XIII. Ed. Academiei R.P.R., Bucuresti: 263-264.
Bardakci, F., Degerli, N., Ozdemir, O., Basibuyuk, H. H. (2006) Phylogeography of the Turkish brown trout Salmo trutta L.: mitochondrial DNA PCR-RFLP variation. Journal of Fish Biology, Volume 68, Supplement 1, March 2006, pp. 36-55(20).
Berg, W.J. and Ferris, S.D., Restriction Endonuclease Analysis of Salmonid Mitochondrial DNA, Can. J. Fish. Aquat. Sci., 1984, vol. 41, pp. 1041-1047.
Bernatchez, L. & Danzmann, R.G. (1993). Congruence in control-region sequence and restriction site variation in mitochondrial DNA of brook charr (Salvelinus fontinalis Mitchill). Molecular Biology and Evolution 10, 1002-1014.
Bernatchez, L. & Osinov, A. (1995). Genetic diversity of trout (genus Salmo) from its most eastern native range based on mitochondrial DNA and nuclear gene variation. Molecular Ecology 4, 285-297.
Bernatchez, L. (2001). The evolutionary history of brown trout (Salmo trutta L.) inferred from phylogeographic, nested clade, and mismatch analysis of mitocondrial DNA variation. Evolution 55: 351-379.
Blanco C. 1995. La trucha cría industrial. Editorial Mundi prensa, Espana, 1-503.
Brown, G., Gadaleta, G., Pepe, G., Saccone, C., Sbisa, E. 1986. Structural conservation and variation in the D-loop containing region of vertebrate mitochondrial DNA. J. Mol. Biol. 192:503-511;
Brown, G., Simpson, M. 1982. Novel features of animals mtDNA evolution as shown of two rat cytochrome oxidase subunitII genes. Proc Nat. Acad. Sci. USA, 79, 3246-50.
Brown, W. M., George M., Wilson A.C., 1979. Rapid evolution of animal mitochondrial DNA, Proc. Natl. Acad. Sci., 76/4, 1967-1971.
Brown, W.M., Prager, E.M., Wang A. 1982. Mitochondrial DNA sequences of primates: tempo and mode of evolution. J. Mol. Evol., 18, 225-39.
Cann, R.L., Brown W.M.,
Carausu, S. 1962. Tratat de Ichtiologie. Ed. Academiei Republicii Populare Romane: 385-391.
Crespi, B.J., Fulton M.J. 2004. Molecular systematics of Salmonidae: combined nuclear data yields a robust phylogeny, Mol. Phylogenet. Evol., 31 (2004) 658-679;
Dixon, M.T., Hillis, D.M., 1993. Ribosomal RNA secondary structure: compensatory mutations and implications for phylogenetic analysis. Mol. Biol. Evol. 10, 256-267.
Doiron S., Blier P.U., Bernatchez L. 1999. NCBI Access N°AF154851.
Doiron S., Blier P.U., Bernatchez L. 2002. NCBI Access N°NC000860.
Domanico, M. J., Phillips, R. B., Oakley, T. H. (1997). Phylogenetic analysis of the Pacific salmon (genus Oncorhynchus) using nuclear and mitochondrial DNA sequences. Can. J. Fish. Aquat. Sci. 54: 1865-1872.
Dorofeyeva, E. A. 1989. The basic principles of classification and phylogeny of the salmonid fishes (Salmoniformes: Salmonoidei: Salmonidae). In ''Biology and Phylogeny of Fishes'' (V. M. Korovinoi, Ed.), pp. 5-16. USSR Academy of Sciences, Proceedings of the Zoological Institute 201, St. Petersburg;
Felsenstein, J., 2004. PHYLIP (Phylogeny Inference Package) version 3.68., Department of Genetics, University of Washington, Seattle;
Ginatulina, L.K., Shedko, S.V., Miroshnichenko, I.L., and Ginatulin, A.A., Divergence of Mitochondrial DNA Sequences in Pacific Salmonids, Zh. Evolyuts. Biokhim.Fiziol., 1988, vol. 24, no. 4, pp. 477-482;
Ginatulina, L.K., Shedko, S.V., Miroshnichenko, I.L., and Ginatulin, A.A., Divergence of Mitochondrial DNA Sequences in Pacific Salmonids, Zh. Evolyuts. Biokhim.Fiziol., 1988, vol. 24, no. 4, pp. 477-482.
Greenberg, B.D., Newbold J.E. and Sugino, A. 1983. Intraspecific nucleotide sequence variability surrounding the origin of replication in human mitochondrial DNA. Gene. 21. 33-49.
Grewe, P.M., Billington, N., and Hebert, P.D.N., Phylogenetic Relationships among Members of Salvelinus Inferred from Mitochondrial DNA Divergence, Can. J. Fish. Aquat. Sci., 1990, vol. 47, pp. 984-991;
Gyllensten, U. and Wilson, A.C., Mitochondrial DNA of Salmonids: Inter-and Intraspecific Variability Detected with Restriction Enzymes, in Population Genetics and Fishery Management, Seattle: Univ. Wash. Press, 1987, pp. 301-318;
Hansen, M.M. and Loeschcke, V. (1996). Genetic differentiation among Danish brown trout (Salmo trutta L.) populations, as detected by RFLP analysis of PCR amplified mitochondrial DNA segments. Journal of Fish Biology, 48, 422-436.
Hedges, S. B. 2002. The origin and evolution of model organisms. Nature Reviews Genetics 3:838-849;
Hixson, J.E., Wong, T.W., Clayton, D.A., 1986. Both the conserved stem-loop and divergent 5V-flanking sequences are required for initiation at the human mitochondrial origin of light-strand DNA-replication. J. Biol. Chem. 261, 2384-2390.
Hurst C.D., Bartlett S.E., Davidson W.S., Bruce I.J., 1999. The complete nucleotide sequence of the mitochondrial DNA of the Atlantic salmon, Salmo salar. Gene 239: 237-242.
Jow, H., Hudelot, C., Rattray, M., Higgs, P. G. 2002. Bayesian phylogenetics using an RNA substitution model applied to early mammalian evolution. Mol. Biol. Evol. 19: 1591-1601.
Kendall, A.W.,Jr., Behnke, R.J. 1984. Salmonidae: Development and relationships. In ''Ontogeny and Systematics of Fishes'' (H. G.Moser, Ed.), pp. 142-149. Am. Soc. of Icthyol. Herpetol. Spec.Publ. 1.
Kitano, T., Matsuoka, N., and Saitou, N., Phylogenetic Relationship of the Genus Oncorhynchus Species Inferred from Nuclear and Mitochondrial Markers, Genes Genet. Syst. 1997, vol. 72, pp. 25-34;
Kottelat, M., Freyhof J. 2007. Handbook of European freshwater fishes. Kottelat, Cornol, Switzerland, Freyhof, Berlin, Germany: 429-430.
Meyer, A. and A.C. Wilson. 1990. Origin of tetrapods inferred from their mitochondrial DNA affiliation to lungfish. J. Mol. Evol. 31: 359-364;
Meyer, A., Dolven, S.I., 1992. Molecules, fossils, and the origin of tetrapods. J. Mol. Evol. 35 (2), 102-113, Review;
Miya M., Nishida M. 2000. Use of Mitogenomic Information in Teleostean Molecular Phylogenetics: A Tree-Based Exploration under the Maximum- Parsimony Optimality Criterion. Mol Phylogenet Evol 17: 437-455;
Miya, M., Nishida, M., 1998. Molecular phylogeny and evolution of the deep-sea fish genus Sternoptyx. Mol. Phylogenet. Evol. 10, 11-22;
Neave F. 1958. The origin and speciation of Oncorhynchus. Trans R. Soc. Can. 551:25-39.
Nikandrov, V.Y., Shindavina, N.I. 2007. Characteristics of the hatchery-reared Black Sea Salmon (Salmo trutta labrax). Journal of Ichthyology, 47(2), 184-193.
Norden, A. 1961. Comparative osteology of representative salmonid fishes, with particular reference to the grayling (Thymallus arcticus) and its phylogeny. J. Fish. Res. Board Can. 8: 679-791.
Oakley T. H., Phillips R.B. Phylogeny of Salmonine Fishes Based on Growth Hormone Introns: Atlantic (Salmo) and Pacific (Oncorhynchus) Salmon Are Not Sister Taxa. Molecular Phylogenetics and Evolution Vol. 11, No. 3, April, pp. 381-393, 1999.
Ohara, I., Sawano, K., and Okazaki, T., Mitochondrial DNA Sequence Analysis of the Masu Salmon-Phylogeny in the Genus Oncorhynchus, Mol. Phylogenet. Evol., 1997, vol. 7, pp. 71-78.
Otel V. 2007. Atlasul pestilor din Rezervatia Biosferei Delta Dunarii. Centrul de Informare Tehnologica Delta Dunarii, Tulcea: 145 - 147.
Otel, V. 2000. Lista Rosie a speciilor de plante si animale din Rezervatia Biosferei Delta Dunarii Romania. Fundatia Aves: 84.
Phillips R.B., Oakley T.H. 1997. Phylogenetic relationships among the Salmonidae based on nuclear DNA and mitochondrial DNA sequences. In: Kocher T, Stepien C. Molecular Systematics of Fishes. San Diego: Academic Press. pp:145-162.
Phillips, R.B., Sajdak, S.L., and Domanico, M.J., Relationships among Chars Based on DNA Sequences, Nordic J. Fresh. Res., 1995, vol. 71, pp. 378-391.
Popescu-Gorj, A., Dumitriu, M. 1956. Somonul Marii Negre (Salmo trutta labrax Pall) migrator in Dunare si baltile inundabile.Bul. Inst. Cerc. Pisc., 15, 4, 31-37.
Radu, Ghe., Radu, E. 2008a. Atlas al principalelor specii de pesti din Marea Neagra. Editura Virom, Constanta: 50-51.
Radu, Ghe., Radu, E. 2008b. Determinator al principalelor specii de pesti din Marea Neagra. Editura Virom, Constanta: 85-86.
Rosenberg, M.S., Kumar, S., 2003. Taxon sampling, bioinformatics, and phylogenomics. Syst. Biol. 52, 119-124.
Saccone, C., Attimonelli, M., and Sbisa, E. 1987. Structural elements highly preserved during the evolution of the D-loop containing region in vertebrate mitochondrial DNA. J. Mol. Evol. 26: 205-211.
Shadel G.S., Clayton D.A. 1997. Mitochondrial DNA maintenance in vertebrates. Ann Rev Biochem 66: 409-435.
Shedlock, A.M., Parker, J.D., Crispin, D.A., Evolution of the Salmonid Mitochondrial Control Region, Mol. Phylogenet. Evol., 1992, vol. 1, pp. 179-192.
Stearley, R.F. 1992. Historical ecology of Salmoninae, with specia reference to Oncorhynchus. In ''Systematics, Historical Ecology, and North American Freshwater Fishes'' (R. L. Mayden, Ed.), Stanford Univ. Press, Stanford, CA.
Stearley, R.F., and Smith, G.R. 1993. Phylogeny of the Pacific trouts and salmons (Oncorhynchus) and genera of the family Salmonidae. Trans. Am. Fish. Soc. 122: 1-33.
Svetovidov, A.N. 1964. Pestii din Marea Neagra (rus). Izdatelstvo Naulea, Moskva si Leningrad: 143-146.
Swofford,
D. L., Olsen, G. J., Waddell, P. J., and Hillis, D. M. 1996. Phylogenetic inference. In: Molecular Systematics,
2nd ed., D. M. Hillis, C. Moritz, and B. K. Mable, eds., pp.407-514. Sinauer
Assoc.,
Tchernavin V. 1939. The origin of salmon. Salmon Trout Mag 95:1-21.
Telford, M.J., Wise, M.J., Gowri-Shankar, V., 2005. Consideration of RNA secondary structure significantly improves likelihood-based estimates of phylogeny: examples from the Bilateria. Mol. Biol. Evol. 22, 1129-1136.
Tiano, T. J., Willis, C. M., Noble, A. A., Luttenton, M. R., Nikitin, A. G. (2007). Genetic identification of hatchery stocks of brown trout using mitochondrial DNA polymorphism. North American Journal of Fisheries Management, 27(3): 965-970.
Thomas, W.K., Beckenbach, A.T. 1989. Variation in Salmonid Mitochondrial DNA: Evolutionary Constrains and Mechanisms of Substitution, J. Mol. Evol., vol. 29, p. 233-245;
Thomas, W.K., Withler, R.E., and Beckenbach, A.T., Mitochondrial DNA Analysis of Pacific Salmonid Evolution, Can. J. Zool., 1986, vol. 64, pp. 1058-1064;
Utter F., Allendorf F. 1994. Phylogenetic relationships among species of Oncorhynchus: a consensus view. Conserv Biol, 8:864-867;
Wolstenholme D.R. 1992. Animal mitochondrial DNA: structure and evolution. Int Rev Cytol 141:173-216.
Wolstenholme, D.R. and Clary, D.O. (1985). Sequence evolution of Drosophilla mitochondrial DNA. Genetics, 109, 725-44.
Zardoya R., Garrido-Pertierra A., Bautista J.M. 1995. The complete nucleotide sequence of the mitochondrial DNA genome of the rainbow trout, Oncorhynchus mykiss. J Mol Evol 41: 942-951.
ANEXA I
Harti privind distributia geografica a salmonidelor pe bazine hidrografice
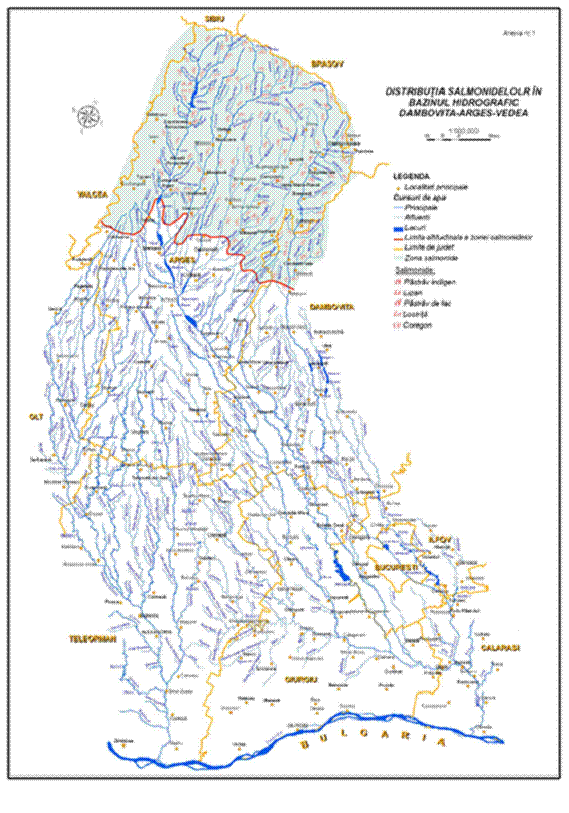
Distributia salmonidelor in bazinul hidrografic Dambovita-Arges-Vedea
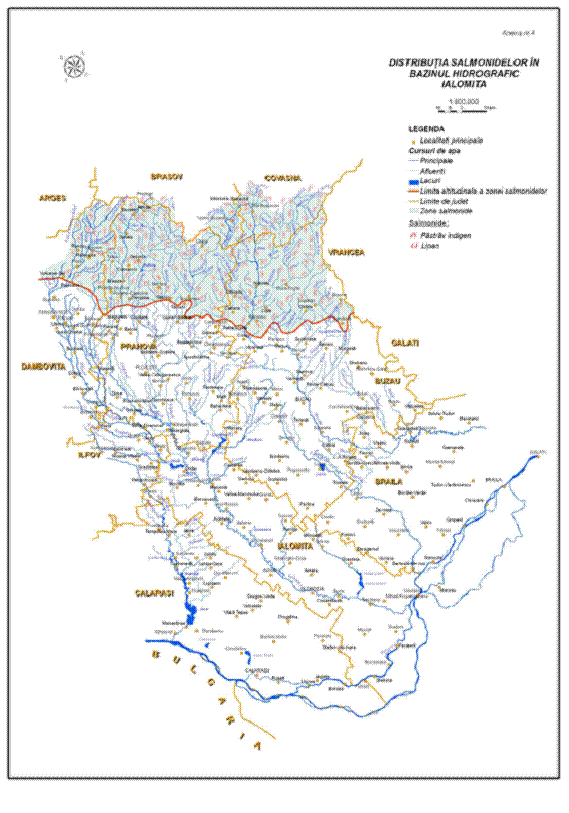
Distributia salmonidelor in bazinul hidrografic Ialomita
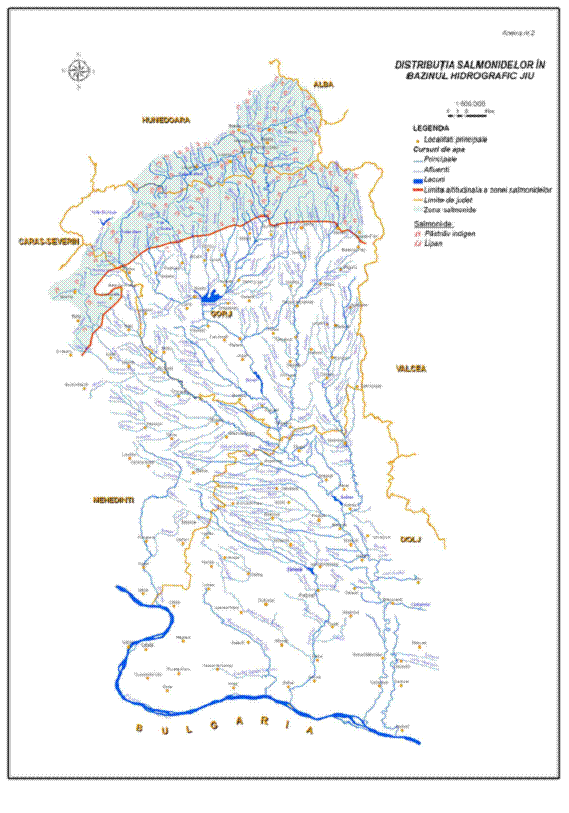
Distributia salmonidelor in bazinul hidrografic Jiu
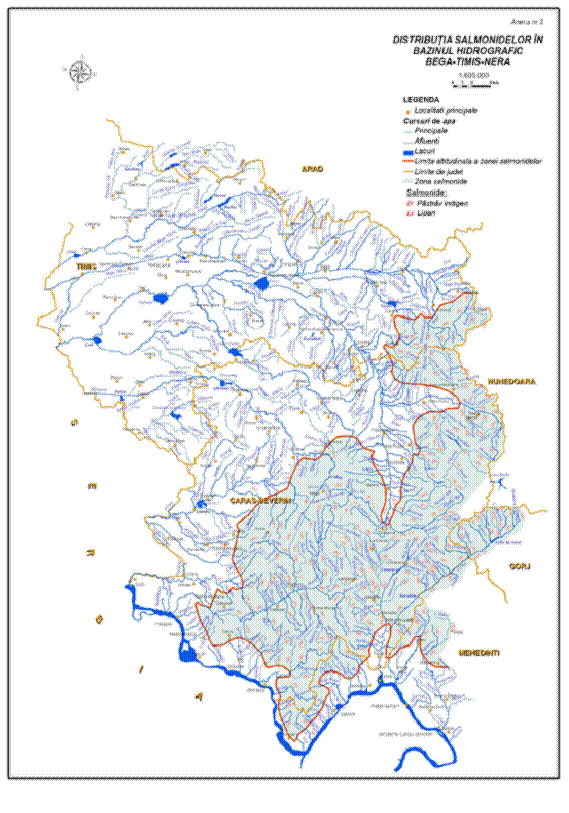
Distributia salmonidelor in bazinul hidrografic Bega-Timis-Nera
Copyright © 2025 - Toate drepturile rezervate
| Hidrologie | ||||
|
||||
|
| ||||
|
| ||||
|
|
||||